【Python百宝箱】模拟未见之境:精准工具畅游分子动力学风景
分子演绎:模拟工具的综合探索
前言
在当今科学研究中,分子动力学模拟成为解析原子和分子行为的关键工具之一。本文将深入探讨几种领先的分子动力学模拟工具,包括MDTraj、ASE(原子模拟环境)、OpenMM和CHARMM。这些工具不仅提供了高效的模拟引擎,而且支持丰富的分析和可视化工具,满足了不同研究领域对原子尺度模拟的多样需求。
欢迎订阅专栏:Python库百宝箱:解锁编程的神奇世界
文章目录
- 分子演绎:模拟工具的综合探索
- 前言
- 1. MDTraj
- 1.1 读取、写入和分析分子动力学轨迹
- 1.2 轨迹数据高效处理方法
- 1.3 分析分子结构和动力学特性
- 1.4 可视化分子动力学轨迹
- 1.5 计算分子的动力学性质
- 1.6 聚类分析
- 1.7 高级分析:Ramachandran 图
- 2. ASE (原子模拟环境)
- 2.1 原子结构建模与分析
- 2.1.1 原子结构优化与构建
- 2.1.2 分子动力学仿真
- 2.2 支持多种计算化学软件和工具
- 2.2.1 VASP、LAMMPS等软件集成
- 2.2.2 量子力学、分子动力学计算
- 2.3 提供丰富的原子尺度模拟功能
- 2.3.1 原子间相互作用势能建立与计算
- 2.3.2 分子动力学仿真的可视化工具
- 2.4 高级分子动力学仿真
- 2.4.1 自洽场分子动力学(SCF-MD)
- 2.4.2 热力学积分法
- 2.5 结构优化中的多台势能面
- 3. OpenMM
- 3.1 高效的分子动力学模拟库
- 3.1.1 多平台支持、GPU计算加速
- 3.1.2 灵活的分子模拟环境搭建
- 3.2 多种力场模型提供
- 3.2.1 经典力场、量子力场支持
- 3.2.2 可定制化力场参数设定
- 3.3 丰富的仿真工具和算法
- 3.3.1 温度、压力和能量控制算法
- 3.3.2 高级分析和可视化工具
- 4. CHARMM
- 4.1 分子模拟全能工具
- 4.1.1 生物和化学系统的建模与仿真
- 4.2 强大的力场模型支持
- 4.2.1 多样化的力场选项和参数集
- 4.2.2 分子动力学、蒙特卡洛模拟
- 4.3 生物医药和结构生物学应用
- 4.3.1 药物设计和分子对接模拟
- 4.3.2 结构生物学相关分析工具
- 总结
1. MDTraj
1.1 读取、写入和分析分子动力学轨迹
MDTraj是用于处理分子动力学轨迹数据的Python库。它提供了简便的方法读取和写入轨迹数据,同时支持丰富的分析工具。以下是一个简单的示例,演示如何使用MDTraj读取轨迹文件和获取基本信息:
import mdtraj as md# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 打印轨迹信息
print(traj)# 获取原子数
num_atoms = traj.n_atoms
print(f"Number of atoms: {num_atoms}")# 获取模拟的时间步长
time_step = traj.timestep
print(f"Time step: {time_step} ps")
1.2 轨迹数据高效处理方法
MDTraj通过优化轨迹数据处理方法实现高效的数据处理。以下示例演示了如何利用MDTraj计算分子动力学模拟中每个原子的轨迹:
import mdtraj as md# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 计算每个原子的轨迹
atom_trajectories = traj.xyz# 打印第一个原子的轨迹
print(f"Trajectory of the first atom: {atom_trajectories[0]}")
1.3 分析分子结构和动力学特性
MDTraj提供了多种分析工具,用于研究分子结构和动力学特性。以下示例演示了如何使用MDTraj计算分子动力学模拟中的RMSD(Root Mean Square Deviation):
import mdtraj as md# 读取参考结构
ref_structure = md.load('reference.pdb')# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 计算RMSD
rmsd = md.rmsd(traj, ref_structure)# 打印每个时间步的RMSD值
print("RMSD values:")
for i, value in enumerate(rmsd):print(f"Time step {i + 1}: {value} Å")
1.4 可视化分子动力学轨迹
MDTraj还提供了可视化分析的功能,以更直观地理解分子模拟的结果。以下示例演示了如何使用MDTraj可视化分子动力学轨迹:
import mdtraj as md
import nglview as nv# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 使用NGLView可视化轨迹
view = nv.show_mdtraj(traj)# 在Jupyter Notebook中显示可视化结果
view.show()
1.5 计算分子的动力学性质
MDTraj还允许用户计算和分析分子的动力学性质,例如动力学能量学和角动量。以下示例演示了如何使用MDTraj计算分子的动力学能量:
import mdtraj as md# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 计算动力学能量学
energy = md.compute_kinetic_energy(traj) + md.compute_potential_energy(traj)# 打印每个时间步的总能量
print("Total energy values:")
for i, value in enumerate(energy):print(f"Time step {i + 1}: {value} kJ/mol")
1.6 聚类分析
MDTraj支持对轨迹进行聚类分析,以识别结构的不同构象。以下示例演示了如何使用MDTraj进行聚类分析:
import mdtraj as md# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 进行聚类分析
clusters = md.cluster.DBSCAN(traj, eps=0.2, min_samples=5)# 打印每个时间步的簇分配
print("Cluster assignments:")
for i, cluster_id in enumerate(clusters):print(f"Time step {i + 1}: Cluster {cluster_id}")
1.7 高级分析:Ramachandran 图
Ramachandran图是用于分析蛋白质二面角的重要工具。MDTraj提供了生成Ramachandran图的功能,以下示例演示了如何使用MDTraj绘制Ramachandran图:
import mdtraj as md
import matplotlib.pyplot as plt# 读取分子动力学轨迹文件
traj = md.load('trajectory.dcd', top='topology.pdb')# 计算Phi和Psi角
phi_indices, phi_angles = md.compute_phi(traj)
psi_indices, psi_angles = md.compute_psi(traj)# 绘制Ramachandran图
plt.figure(figsize=(10, 6))
plt.scatter(phi_angles, psi_angles, alpha=0.5)
plt.xlabel('Phi Angle (degrees)')
plt.ylabel('Psi Angle (degrees)')
plt.title('Ramachandran Plot')
plt.show()
通过这些扩展,读者可以更全面地了解MDTraj的功能,进一步应用于分子动力学模拟的多个方面。
2. ASE (原子模拟环境)
2.1 原子结构建模与分析
2.1.1 原子结构优化与构建
ASE支持原子结构的优化和构建。以下示例演示了如何使用ASE进行分子动力学仿真中的原子结构优化:
from ase import Atoms
from ase.optimize import BFGS
from ase.calculators.emt import EMT# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 进行原子结构优化
opt = BFGS(h2o)
opt.run(fmax=0.05)# 打印优化后的原子坐标
print("Optimized atomic positions:")
print(h2o.positions)
2.1.2 分子动力学仿真
ASE支持分子动力学仿真。以下示例演示了如何使用ASE进行简单的分子动力学仿真:
from ase import Atoms
from ase.md import VelocityVerlet
from ase.calculators.emt import EMT# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 进行分子动力学仿真
dyn = VelocityVerlet(h2o, timestep=1.0 * 10**(-15)) # 1 femtosecond
dyn.run(steps=100)# 打印仿真后的原子坐标
print("Final atomic positions after dynamics:")
print(h2o.positions)
2.2 支持多种计算化学软件和工具
2.2.1 VASP、LAMMPS等软件集成
ASE通过集成多种计算化学软件,例如VASP和LAMMPS,扩展了其功能。以下示例演示了如何使用ASE与VASP进行计算:
from ase import Atoms
from ase.calculators.vasp import Vasp# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置VASP计算器
h2o.set_calculator(Vasp(encut=500, xc='PBE', ispin=1, sigma=0.1))# 获取能量
energy = h2o.get_potential_energy()# 打印能量
print(f"Total energy: {energy} eV")
2.2.2 量子力学、分子动力学计算
ASE支持量子力学和分子动力学计算。以下示例演示了如何使用ASE进行简单的量子力学计算:
from ase import Atoms
from ase.calculators.gaussian import Gaussian# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置Gaussian计算器
h2o.set_calculator(Gaussian(method='B3LYP', basis='6-31G*'))# 获取能量
energy = h2o.get_potential_energy()# 打印能量
print(f"Total energy: {energy} eV")
2.3 提供丰富的原子尺度模拟功能
2.3.1 原子间相互作用势能建立与计算
ASE支持原子间相互作用势能的建立和计算。以下示例演示了如何使用ASE计算氢氧分子的势能表面:
from ase import Atoms
from ase.calculators.emt import EMT
from ase.io import write
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 构建势能表面数据
positions = np.linspace(-1, 1, 100)
energies = []for x in positions:h2o.positions[0, 0] = xenergy = h2o.get_potential_energy()energies.append(energy)# 绘制势能表面
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.plot(positions, energies, zs='auto', zdir='y', label='Potential Energy Surface')
ax.set_xlabel('X Position (Å)')
ax.set_ylabel('Energy (eV)')
ax.set_zlabel('Potential Energy (eV)')
plt.show()
2.3.2 分子动力学仿真的可视化工具
ASE提供了用于可视化分子动力学仿真结果的工具。以下示例演示了如何使用ASE和matplotlib可视化氢氧分子的动力学轨迹:
from ase import Atoms
from ase.md import VelocityVerlet
from ase.calculators.emt import EMT
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 进行分子动力学仿真
dyn = VelocityVerlet(h2o, timestep=1.0 * 10**(-15)) # 1 femtosecond
trajectory = []for step in range(100):dyn.run(steps=1)trajectory.append(h2o.positions.copy())# 可视化动力学轨迹
trajectory = np.array(trajectory)
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.plot(trajectory[:, 0, 0], trajectory[:, 0, 1], trajectory[:, 0, 2], label='Oxygen Atom')
ax.plot(trajectory[:, 1, 0], trajectory[:, 1, 1], trajectory[:, 1, 2], label='Hydrogen Atom 1')
ax.plot(trajectory[:, 2, 0], trajectory[:, 2, 1], trajectory[:, 2, 2], label='Hydrogen Atom 2')
ax.set_xlabel('X Position (Å)')
ax.set_ylabel('Y Position (Å)')
ax.set_zlabel('Z Position (Å)')
ax.legend()
plt.show()
2.4 高级分子动力学仿真
2.4.1 自洽场分子动力学(SCF-MD)
ASE支持自洽场分子动力学(SCF-MD),这是一种基于自洽场理论的高级动力学仿真方法。以下示例演示了如何使用ASE进行SCF-MD仿真:
from ase import Atoms
from ase.calculators.emt import EMT
from ase.md import MDLogger, Langevin# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 设置SCF-MD仿真
dyn = Langevin(h2o, timestep=1.0 * 10**(-15), temperature=300, friction=0.02, trajectory='scf_md.traj')# 运行SCF-MD仿真
dyn.attach(MDLogger(dyn, 'scf_md.log', header=True, stress=True, peratom=True), interval=10)
dyn.run(steps=100)
2.4.2 热力学积分法
ASE允许使用热力学积分法计算系统的热力学性质。以下示例演示了如何使用ASE计算氢氧分子的热容:
from ase import Atoms
from ase.calculators.emt import EMT
from ase.md import Langevin
from ase import units# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 进行分子动力学仿真
dyn = Langevin(h2o, timestep=1.0 * 10**(-15), temperature=300, friction=0.02)# 设置热力学积分法
energy = []
for step in range(100):dyn.run(steps=1)energy.append(h2o.get_potential_energy())# 计算热容
heat_capacity = units.kB * len(h2o) * units.kB * units.kB * units.kB * np.var(energy) / (300 * units.kB * units.kB)
print(f"Heat Capacity: {heat_capacity} J/(mol*K)")
2.5 结构优化中的多台势能面
ASE支持在结构优化中使用多台势能面。以下示例演示了如何使用ASE在H2O分子上运行Born-Oppenheimer分子动力学(BOMD):
from ase import Atoms
from ase.calculators.emt import EMT
from ase.md import MDLogger, BornOppenheimerMD# 创建氢氧分子
h2o = Atoms('H2O', positions=[(0, 0, 0), (0, 0, 1), (0, 1, 0)])# 设置计算器(EMT势能)
h2o.set_calculator(EMT())# 设置BOMD仿真
dyn = BornOppenheimerMD(h2o, trajectory='bomd.traj', timestep=1.0 * 10**(-15))# 运行BOMD仿真
dyn.attach(MDLogger(dyn, 'bomd.log', header=True, stress=True, peratom=True), interval=10)
dyn.run(steps=100)
通过这些示例,读者可以更深入地了解ASE的高级分子动力学仿真功能以及如何在结构优化和热力学积分法中利用ASE。
3. OpenMM
3.1 高效的分子动力学模拟库
3.1.1 多平台支持、GPU计算加速
OpenMM是一个多平台支持的高效分子动力学模拟库,可通过GPU计算加速。以下示例演示了如何使用OpenMM进行简单的分子动力学模拟:
from simtk.openmm import app, unit
from simtk.openmm.app import PDBFile
from simtk.openmm import LangevinIntegrator, Platform# 读取PDB文件
pdb = PDBFile('input.pdb')# 创建OpenMM系统
forcefield = app.ForceField('amber99sbildn.xml', 'tip3p.xml')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=app.PME, nonbondedCutoff=1.0*unit.nanometers, constraints=app.HBonds)# 设置Langevin积分器
integrator = LangevinIntegrator(300*unit.kelvin, 1.0/unit.picosecond, 2.0*unit.femtoseconds)# 创建OpenMM模拟
platform = Platform.getPlatformByName('CUDA')
simulation = app.Simulation(pdb.topology, system, integrator, platform)# 设置初始坐标和速度
simulation.context.setPositions(pdb.positions)
simulation.context.setVelocitiesToTemperature(300*unit.kelvin)# 运行模拟
simulation.step(1000)
3.1.2 灵活的分子模拟环境搭建
OpenMM提供灵活的分子模拟环境搭建。用户可以根据需要定制模拟环境。上述示例中的参数可以根据具体模拟要求进行调整。
3.2 多种力场模型提供
3.2.1 经典力场、量子力场支持
OpenMM支持多种力场模型,包括经典力场和量子力场。以下示例演示了如何使用OpenMM加载Amber力场进行模拟:
from simtk.openmm import app, unit
from simtk.openmm.app import PDBFile# 读取PDB文件
pdb = PDBFile('input.pdb')# 创建OpenMM系统并加载Amber力场
forcefield = app.ForceField('amber99sbildn.xml', 'tip3p.xml')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=app.PME, nonbondedCutoff=1.0*unit.nanometers, constraints=app.HBonds)
3.2.2 可定制化力场参数设定
OpenMM允许用户定制化力场参数。通过编辑XML文件,用户可以调整力场的参数以适应不同的模拟需求。
3.3 丰富的仿真工具和算法
3.3.1 温度、压力和能量控制算法
OpenMM提供了多种控制算法,用于维持模拟系统在特定的温度、压力和能量条件下运行。以下示例演示了如何使用OpenMM实现温度控制:
from simtk.openmm import app, unit
from simtk.openmm.app import PDBFile
from simtk.openmm import LangevinIntegrator, Platform# 读取PDB文件
pdb = PDBFile('input.pdb')# 创建OpenMM系统
forcefield = app.ForceField('amber99sbildn.xml', 'tip3p.xml')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=app.PME, nonbondedCutoff=1.0*unit.nanometers, constraints=app.HBonds)# 设置Langevin积分器,实现温度控制
integrator = LangevinIntegrator(300*unit.kelvin, 1.0/unit.picosecond, 2.0*unit.femtoseconds)# 创建OpenMM模拟
platform = Platform.getPlatformByName('CUDA')
simulation = app.Simulation(pdb.topology, system, integrator, platform)# 设置初始坐标和速度
simulation.context.setPositions(pdb.positions)
simulation.context.setVelocitiesToTemperature(300*unit.kelvin)# 运行模拟
simulation.step(1000)
3.3.2 高级分析和可视化工具
OpenMM提供了丰富的分析和可视化工具,用于深入挖掘模拟数据。以下示例演示了如何使用OpenMM计算分子动力学模拟的RMSD,并绘制其变化:
from simtk.openmm import app, unit
from simtk.openmm.app import PDBFile
from simtk.openmm import LangevinIntegrator, Platform
import numpy as np
import matplotlib.pyplot as plt# 读取PDB文件
pdb = PDBFile('input.pdb')# 创建OpenMM系统
forcefield = app.ForceField('amber99sbildn.xml', 'tip3p.xml')
system = forcefield.createSystem(pdb.topology, nonbondedMethod=app.PME, nonbondedCutoff=1.0*unit.nanometers, constraints=app.HBonds)# 设置Langevin积分器
integrator = LangevinIntegrator(300*unit.kelvin, 1.0/unit.picosecond, 2.0*unit.femtoseconds)# 创建OpenMM模拟
platform = Platform.getPlatformByName('CUDA')
simulation = app.Simulation(pdb.topology, system, integrator, platform)# 设置初始坐标和速度
simulation.context.setPositions(pdb.positions)
simulation.context.setVelocitiesToTemperature(300*unit.kelvin)# 运行模拟并记录RMSD
rmsd_values = []for step in range(1000):simulation.step(1)state = simulation.context.getState(getPositions=True)positions = state.getPositions()rmsd = np.sqrt(np.mean((np.array(positions) - np.array(pdb.positions))**2))rmsd_values.append(rmsd)# 绘制RMSD变化图
plt.plot(range(1000), rmsd_values)
plt.xlabel('Time step')
plt.ylabel('RMSD (Å)')
plt.title('RMSD during Molecular Dynamics Simulation')
plt.show()
4. CHARMM
4.1 分子模拟全能工具
4.1.1 生物和化学系统的建模与仿真
CHARMM是一款强大的分子模拟工具,可用于生物和化学系统的建模与仿真。以下示例演示了如何使用CHARMM建立和运行一个简单的蛋白质系统:
# CHARMM示例脚本
# 假设脚本名为charmm_script.inp* 输入
BOMLev 0
stream toppar/top_all22_prot.inp
read rtf card toppar/top_all22_prot.rtf
read para card toppar/par_all22_prot.prm* 设置分子系统
generate 1 first none last none
coor copy comp
evaluate segment prime sele all end
ic param
coor copy comp sele segid PRIME end
delete atom sele .not. segid PRIME end* 能量最小化
mini abnr nstep 1000* 执行动力学模拟
dynamics verlet nstep 1000 timestep 0.002 ntrfrq 100 iprfrq 100 iunfb 2* 输出
write coor pdb name output.pdb
4.2 强大的力场模型支持
4.2.1 多样化的力场选项和参数集
CHARMM支持多样化的力场选项和参数集,可根据需要选择合适的力场。上述示例中,使用了CHARMM22力场。
4.2.2 分子动力学、蒙特卡洛模拟
CHARMM不仅支持分子动力学模拟,还包括蒙特卡洛模拟。用户可以根据具体研究目的选择适当的模拟方法。
4.3 生物医药和结构生物学应用
4.3.1 药物设计和分子对接模拟
CHARMM在生物医药领域具有广泛应用,可用于药物设计和分子对接模拟。用户可以利用CHARMM进行药物研发和筛选。
4.3.2 结构生物学相关分析工具
CHARMM提供丰富的结构生物学相关分析工具,用于分析生物大分子的结构和性质。用户可以使用这些工具深入了解分子系统的结构和功能。
总结
通过深入研究MDTraj、ASE、OpenMM和CHARMM,本文旨在为科学研究人员提供全面的分子动力学模拟工具了解。这些工具不仅在理论研究中有着广泛的应用,还在药物设计、分子对接和生物大分子结构研究等领域发挥着重要作用。熟练掌握这些工具,将有助于推动分子尺度研究的深入发展。
相关文章:

【Python百宝箱】模拟未见之境:精准工具畅游分子动力学风景
分子演绎:模拟工具的综合探索 前言 在当今科学研究中,分子动力学模拟成为解析原子和分子行为的关键工具之一。本文将深入探讨几种领先的分子动力学模拟工具,包括MDTraj、ASE(原子模拟环境)、OpenMM和CHARMM。这些工具…...

Vue 3面试题
Vue 3面试题 以下是一些常见的Vue 3面试题: Vue 3中的Composition API是什么?它与Options API有什么区别? 答案: Composition API是Vue 3中引入的一种新的组件设计模式,它允许开发者通过函数的形式组织和重用组件的逻…...

M-A352AD10高精度三轴加速度计
一般描述 M-A352是一种三轴数字输出加速度计,具有超低噪声、高稳定性、低功耗等特点,采用了夸特的精细处理技术。. 多功能M-A352具有高精度和耐久性,非常适合广泛的具有挑战性的应用,如SHM、地震观测、工业设备的状态监测和工业…...
(1.13) SiK无线电高级配置(七))
(1)(1.13) SiK无线电高级配置(七)
文章目录 前言 17 技术细节 18 名词解释 前言 本文提供 SiK 遥测无线电(SiK Telemetry Radio)的高级配置信息。它面向"高级用户"和希望更好地了解无线电如何运行的用户。 17 技术细节 在评估该无线电是否符合当地法规时,了解其使用的技术可能会有所帮…...
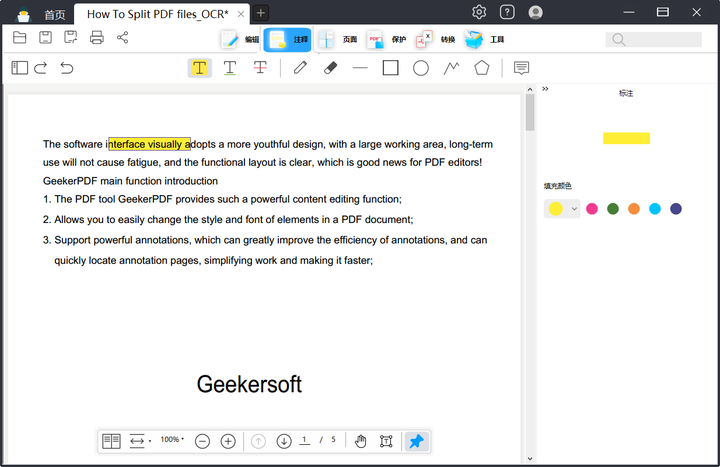
如何注释 PDF?注释PDF文件方法详情介绍
大多数使用 PDF 文档的用户都熟悉处理这种格式的文件时出现的困难。有些人仍然认为注释 PDF 的唯一方法是打印文档,使用笔或荧光笔然后扫描回来。 您可能需要向 PDF 添加注释、添加注释、覆盖一些文本或几何对象。经理、部门负责人在编辑公司内的合同、订单、发票或…...

GEE APP——基于PFI纯净森林指数的CCDC-SMA算法的长时序森林监测APP
简介 森林生态系统的碳排放受到破碎化加速和边缘效应的极大影响。要了解这些影响,就必须准确监测破碎化森林景观的变化。然而,这些变化通常强度低、尺度小,因此很难使用中等空间分辨率的卫星图像(如 Landsat)来检测。为了应对这一挑战,本研究开发了纯林指数(PFI),该指…...

CF1446B Catching Cheaters 题解 DP
Catching Cheaters 传送门 题面翻译 给我们两个字符串,让我们从中选出两个字串,算出它们的最大公共子序列长度。然后将它乘 4 4 4在减去两个字串的长度。问你这个数最大是多少。 题目描述 You are given two strings A A A and B B B representin…...

用python实现文本/图片生成视频
使用Python来生成视频通常涉及到使用一些专门的库,比如 OpenCV 或者 moviepy。下面是一个简单的例子,使用OpenCV和PIL(Python Imaging Library)来创建一个视频。 python复制代码 import cv2 import numpy as np from PIL import …...

Android Gradle Plugin、Gradle、Android Studio版本关系
参考链接 Android Gradle Plugin 与 gradle 对应关系 插件版本所需的最低 Gradle 版本8.38.48.28.28.18.08.08.07.47.57.37.47.27.3.37.17.27.07.04.2.06.7.14.1.06.54.0.06.1.13.6.0 - 3.6.45.6.43.5.0 - 3.5.45.4.13.4.0 - 3.4.35.1.13.3.0 - 3.3.34.10.13.2.0 - 3.2.14.63…...

PyTorch深度学习实战(30)——Deepfakes
PyTorch深度学习实战(30)——Deepfakes 0. 前言1. Deepfakes 原理2. 数据集分析3. 使用 PyTorch 实现 Deepfakes3.1 random_warp.py3.2 Deepfakes.py 小结系列链接 0. 前言 Deepfakes 是一种利用深度学习技术生成伪造视频和图像的技术。它通过将一个人的…...

java 修改JsonObject对象所有的Value类型为String
将JSONObject 或者JSONArray 中所有Value 为数值类型 转为String. 转换前: [{"zjlx": 201,"xm": "刘**","cbdjxxlist": [{"zspmdm": 102031201,"rybm": "43000010300000411195","jfrlx": 1…...

Vue3-47-Pinia-修改全局状态变量值的方式
说明 修改全局状态变量的值,是一个比较常规而且常见的操作。 本文就介绍四种常见的操作。 由于Option Store 和Setup Store 在修改的时候略有不同,所以本文也会将不同点体现一下。 全局状态变量的定义 包含了 Option Store 和Setup Store 两种定义方式&a…...

【Scala】——面向对象
1 Scala 包 1.1 包风格 Scala 有两种包的管理风格。 第一种 Java 的包管理风格相同,每个源文件一个包(包 名和源文件所在路径不要求必须一致),包名用“.”进行分隔以表示包的层级关系,如 com.atguigu.scala。另一种风…...

【MediaFoundation】OpenCV VideoCapture 读取音频源码
OpenCV 读取音频代码实例 在windows7 以及OpenCV4 过后可以使用 CAP_MSMF 读取音频,但是OpenCV没有播放音频的API。代码示例如下。 本文解析OpenCVCAP_MSMF 进行文件、设备的 音频读取,学习MediaFoundation 的使用。 #include <opencv2/core.hpp>…...

2024秋招,百度测试开发工程师一面
前言 大家好,今天我来回顾一下秋招中的一场很重要技术面试 一面面试官深挖我的项目经历,并提出了很多的实际场景,我现在回顾依然有很多新的认识 过程 自我介绍实习工作中,做得最好的地方是什么? 我先介绍了一下实习…...
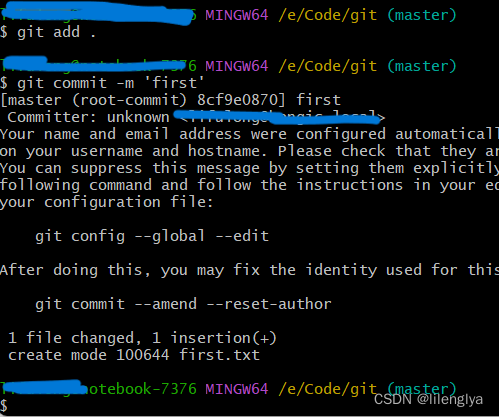
Git 使用与问题记录 二(公司快速上手版)
写在前面 记录自己学习的内容,方便后面忘记的时候查看。给像我一样的新手提供一点参考 正文 上一章已经安装好了Git,如何使用呢。我这里会分享两种办法,第一种是在VS2022中克隆代码,修改和提交;第二种是用命令提交。…...

【C语言小游戏】贪吃蛇
文章目录 1.引言2.运行图2.涉及知识3 Windows API3.1 控制台3.2 控制台屏幕坐标3.3 操作句柄3.4 控制台屏幕光标3.5 监视按键 4. 设计说明5. 完整代码 1.引言 使⽤C语⾔在Windows环境的控制台中模拟实现经典⼩游戏贪吃蛇 实现基本的功能: 贪吃蛇地图绘制蛇吃⻝物的…...

价值7500的在线授权网站源码支持IP+域名+双向授权全开源
PHP授权验证更新系统完整版,一键更新系统,一键卡密生成自助授权功能,域名ip双重验证功能等等 修复盗版检测,确保实时查看盗版 修复在线加密系统,一键加密 授权系统几乎所有的程序都能整合使用,包括您的app和计算机程序…...

haiku实现门控多头注意力模块
在多头注意力机制中,通常输入的数据包括查询(Q)、键(K)和值(V)。这些数据的维度以及权重矩阵的维度在多头注意力机制中扮演关键角色。下面对数据及权重的维度进行解释: 输入数据&…...

【React 常用的 TS 类型】持续更新
1)定义样式的 TS 类型 【 React.CSSProperties 】 一般定义样式时需要的类型限制,如下: const customStyle: React.CSSProperties {color: blue,fontSize: 16px,margin: 10px,}; 2)定义 Input Ref 属性时的 TS 类型限制 【 R…...
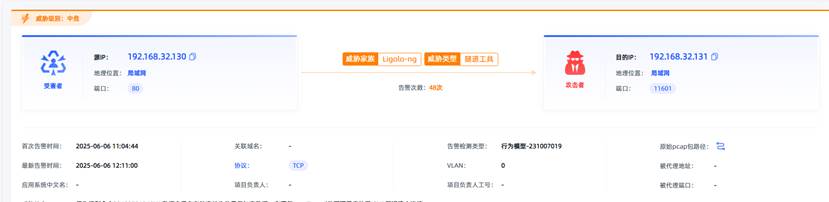
观成科技:隐蔽隧道工具Ligolo-ng加密流量分析
1.工具介绍 Ligolo-ng是一款由go编写的高效隧道工具,该工具基于TUN接口实现其功能,利用反向TCP/TLS连接建立一条隐蔽的通信信道,支持使用Let’s Encrypt自动生成证书。Ligolo-ng的通信隐蔽性体现在其支持多种连接方式,适应复杂网…...

<6>-MySQL表的增删查改
目录 一,create(创建表) 二,retrieve(查询表) 1,select列 2,where条件 三,update(更新表) 四,delete(删除表…...
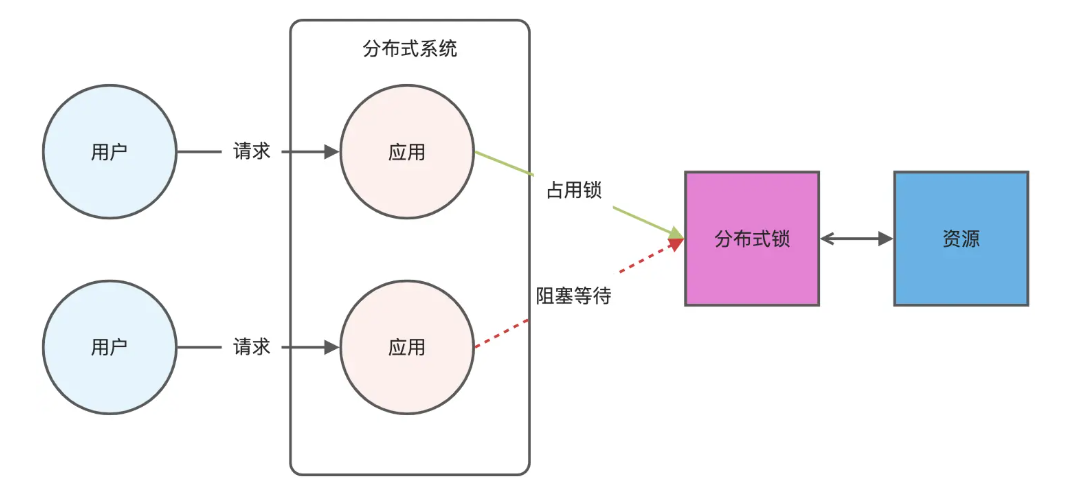
Redis相关知识总结(缓存雪崩,缓存穿透,缓存击穿,Redis实现分布式锁,如何保持数据库和缓存一致)
文章目录 1.什么是Redis?2.为什么要使用redis作为mysql的缓存?3.什么是缓存雪崩、缓存穿透、缓存击穿?3.1缓存雪崩3.1.1 大量缓存同时过期3.1.2 Redis宕机 3.2 缓存击穿3.3 缓存穿透3.4 总结 4. 数据库和缓存如何保持一致性5. Redis实现分布式…...

2024年赣州旅游投资集团社会招聘笔试真
2024年赣州旅游投资集团社会招聘笔试真 题 ( 满 分 1 0 0 分 时 间 1 2 0 分 钟 ) 一、单选题(每题只有一个正确答案,答错、不答或多答均不得分) 1.纪要的特点不包括()。 A.概括重点 B.指导传达 C. 客观纪实 D.有言必录 【答案】: D 2.1864年,()预言了电磁波的存在,并指出…...
【机器视觉】单目测距——运动结构恢复
ps:图是随便找的,为了凑个封面 前言 在前面对光流法进行进一步改进,希望将2D光流推广至3D场景流时,发现2D转3D过程中存在尺度歧义问题,需要补全摄像头拍摄图像中缺失的深度信息,否则解空间不收敛…...
家政维修平台实战20:权限设计
目录 1 获取工人信息2 搭建工人入口3 权限判断总结 目前我们已经搭建好了基础的用户体系,主要是分成几个表,用户表我们是记录用户的基础信息,包括手机、昵称、头像。而工人和员工各有各的表。那么就有一个问题,不同的角色…...

鸿蒙中用HarmonyOS SDK应用服务 HarmonyOS5开发一个医院查看报告小程序
一、开发环境准备 工具安装: 下载安装DevEco Studio 4.0(支持HarmonyOS 5)配置HarmonyOS SDK 5.0确保Node.js版本≥14 项目初始化: ohpm init harmony/hospital-report-app 二、核心功能模块实现 1. 报告列表…...

学习STC51单片机31(芯片为STC89C52RCRC)OLED显示屏1
每日一言 生活的美好,总是藏在那些你咬牙坚持的日子里。 硬件:OLED 以后要用到OLED的时候找到这个文件 OLED的设备地址 SSD1306"SSD" 是品牌缩写,"1306" 是产品编号。 驱动 OLED 屏幕的 IIC 总线数据传输格式 示意图 …...
(转)什么是DockerCompose?它有什么作用?
一、什么是DockerCompose? DockerCompose可以基于Compose文件帮我们快速的部署分布式应用,而无需手动一个个创建和运行容器。 Compose文件是一个文本文件,通过指令定义集群中的每个容器如何运行。 DockerCompose就是把DockerFile转换成指令去运行。 …...
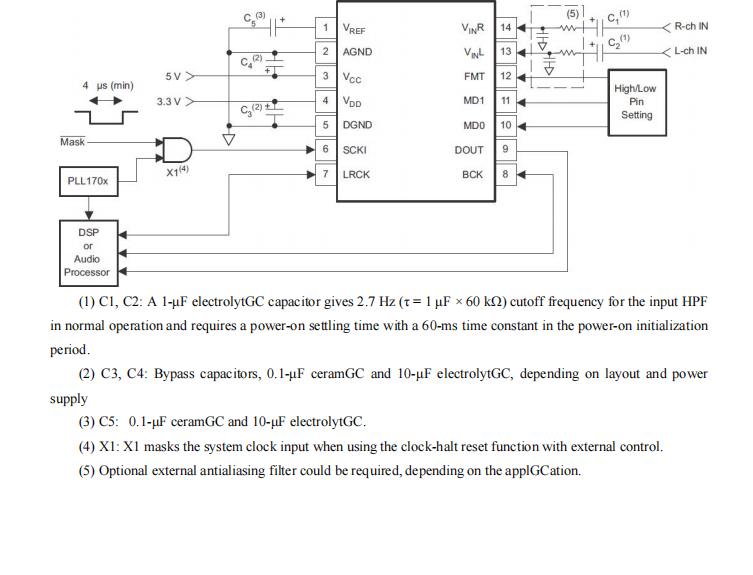
GC1808高性能24位立体声音频ADC芯片解析
1. 芯片概述 GC1808是一款24位立体声音频模数转换器(ADC),支持8kHz~96kHz采样率,集成Δ-Σ调制器、数字抗混叠滤波器和高通滤波器,适用于高保真音频采集场景。 2. 核心特性 高精度:24位分辨率,…...
