单细胞Seruat和h5ad数据格式互换(R与python)方法学习和整理
SeruatV4数据转化为h5ad格式数据
1、导入(R)
rm(list = ls())
library(Seurat)
library(qs)
library(reticulate)
library(hdf5r)
library(sceasy)
library(BiocParallel)
register(MulticoreParam(workers = 4, progressbar = TRUE))
scRNA <- qread("sc_dataset.qs")
scRNA
# An object of class Seurat
# 30269 features across 44651 samples within 2 assays
# Active assay: integrated (2000 features, 2000 variable features)
# 1 other assay present: RNA
# 3 dimensional reductions calculated: pca, umap, tsne
2、配置python环境(终端/linux)
# 配置环境
conda create -n sceasy python=3.9
conda activate sceasy
conda install loompy# 可选安装
conda install anndata
conda install scipy
3、开始转换(R)
# 在R语言中加载python环境
use_condaenv('sceasy')
loompy <- reticulate::import('loompy')# Seurat to AnnData
sceasy::convertFormat(scRNA, from="seurat", to="anndata",outFile='scRNA.h5ad')
# AnnData object with n_obs × n_vars = 44651 × 28269
# obs: 'orig.ident', 'nCount_RNA', 'nFeature_RNA', 'GSE_num', 'Gender', 'Age', 'subsite', 'hpv', 'percent.mt', 'percent.rp', 'percent.hb', 'RNA_snn_res.0.1', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3', 'RNA_snn_res.0.4', 'RNA_snn_res.0.5', 'RNA_snn_res.0.6', 'RNA_snn_res.0.7', 'RNA_snn_res.0.8', 'RNA_snn_res.0.9', 'RNA_snn_res.1', 'RNA_snn_res.1.1', 'RNA_snn_res.1.2', 'RNA_snn_res.1.3', 'RNA_snn_res.1.4', 'RNA_snn_res.1.5', 'RNA_snn_res.1.6', 'RNA_snn_res.1.7', 'RNA_snn_res.1.8', 'RNA_snn_res.1.9', 'RNA_snn_res.2', 'seurat_clusters', 'celltype', 'integrated_snn_res.0.1', 'integrated_snn_res.0.2', 'integrated_snn_res.0.3', 'integrated_snn_res.0.4', 'integrated_snn_res.0.5', 'integrated_snn_res.0.6', 'integrated_snn_res.0.7', 'integrated_snn_res.0.8', 'integrated_snn_res.0.9', 'integrated_snn_res.1', 'integrated_snn_res.1.1', 'integrated_snn_res.1.2', 'integrated_snn_res.1.3', 'integrated_snn_res.1.4', 'integrated_snn_res.1.5', 'integrated_snn_res.1.6', 'integrated_snn_res.1.7', 'integrated_snn_res.1.8', 'integrated_snn_res.1.9', 'integrated_snn_res.2'
# var: 'name'
# obsm: 'X_pca', 'X_umap', 'X_tsne'#Seurat to SingleCellExperiment
sceasy::convertFormat(scRNA, from="seurat", to="sce",outFile='scRNA.rds')
4、IDE中确认一下(python)
# 加载库
import scanpy as sc
import os# 确认路径
os.getcwd()# 读取数据
adata = sc.read_h5ad('scRNA.h5ad')
adata
# AnnData object with n_obs × n_vars = 44651 × 28269
# obs: 'orig.ident', 'nCount_RNA', 'nFeature_RNA', 'GSE_num', 'Gender', 'Age', 'subsite', 'hpv', 'percent.mt', 'percent.rp', 'percent.hb', 'RNA_snn_res.0.1', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3', 'RNA_snn_res.0.4', 'RNA_snn_res.0.5', 'RNA_snn_res.0.6', 'RNA_snn_res.0.7', 'RNA_snn_res.0.8', 'RNA_snn_res.0.9', 'RNA_snn_res.1', 'RNA_snn_res.1.1', 'RNA_snn_res.1.2', 'RNA_snn_res.1.3', 'RNA_snn_res.1.4', 'RNA_snn_res.1.5', 'RNA_snn_res.1.6', 'RNA_snn_res.1.7', 'RNA_snn_res.1.8', 'RNA_snn_res.1.9', 'RNA_snn_res.2', 'seurat_clusters', 'celltype', 'integrated_snn_res.0.1', 'integrated_snn_res.0.2', 'integrated_snn_res.0.3', 'integrated_snn_res.0.4', 'integrated_snn_res.0.5', 'integrated_snn_res.0.6', 'integrated_snn_res.0.7', 'integrated_snn_res.0.8', 'integrated_snn_res.0.9', 'integrated_snn_res.1', 'integrated_snn_res.1.1', 'integrated_snn_res.1.2', 'integrated_snn_res.1.3', 'integrated_snn_res.1.4', 'integrated_snn_res.1.5', 'integrated_snn_res.1.6', 'integrated_snn_res.1.7', 'integrated_snn_res.1.8', 'integrated_snn_res.1.9', 'integrated_snn_res.2'
# var: 'name'
# obsm: 'X_pca', 'X_tsne', 'X_umap'
SeruatV5数据转化为h5ad格式数据
1、导入(R)
rm(list = ls())
V5_path = "/Library/Frameworks/R.framework/Versions/4.4-arm64/Resources/seurat5/"
.libPaths(V5_path)
.libPaths()
library(Seurat)
library(qs)
library(reticulate)
library(hdf5r)
library(sceasy)
library(BiocParallel)
register(MulticoreParam(workers = 4, progressbar = TRUE))
scRNA_V5 <- readRDS("scRNA_tumor.rds")
scRNA_V5
# An object of class Seurat
# 20124 features across 5042 samples within 1 assay
# Active assay: RNA (20124 features, 2000 variable features)
# 3 layers present: counts, data, scale.data
# 3 dimensional reductions calculated: pca, harmony, umap
2、配置python环境(终端/linux)
# 配置环境
conda create -n sceasy python=3.9
conda activate sceasy
conda install loompy# 可选安装
conda install anndata
conda install scipy
3、R语言转换(R)
# 在R语言中加载python环境
use_condaenv('sceasy')
loompy <- reticulate::import('loompy')# Seurat to AnnData
scRNA_V5[["RNA"]] <- as(scRNA_V5[["RNA"]], "Assay")
sceasy::convertFormat(scRNA_V5, from="seurat", to="anndata",outFile='scRNA_V5.h5ad')
# AnnData object with n_obs × n_vars = 5042 × 20124
# obs: 'nCount_RNA', 'nFeature_RNA', 'Sample', 'Cell.Barcode', 'Type', 'RNA_snn_res.0.1', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3', 'RNA_snn_res.0.4', 'RNA_snn_res.0.5', 'RNA_snn_res.0.6', 'RNA_snn_res.0.7', 'RNA_snn_res.0.8', 'RNA_snn_res.0.9', 'RNA_snn_res.1', 'RNA_snn_res.1.1', 'RNA_snn_res.1.2', 'seurat_clusters', 'celltype', 'seurat_annotation'
# var: 'vf_vst_counts_mean', 'vf_vst_counts_variance', 'vf_vst_counts_variance.expected', 'vf_vst_counts_variance.standardized', 'vf_vst_counts_variable', 'vf_vst_counts_rank', 'var.features', 'var.features.rank'
# obsm: 'X_pca', 'X_harmony', 'X_umap'
# Warning message:
# In .regularise_df(obj@meta.data, drop_single_values = drop_single_values) :
# Dropping single category variables:orig.ident
先将 Seurat V5 对象中的 Assay5 类型转换为 Seurat 旧版本中的 Assay 类型,然后再进行转化
4、IDE中确认一下(python)
# 加载库
import scanpy as sc
import os# 确认路径
os.getcwd()# 读取数据
adata = sc.read_h5ad('scRNA.h5ad')
adata
# AnnData object with n_obs × n_vars = 5042 × 20124
# obs: 'nCount_RNA', 'nFeature_RNA', 'Sample', 'Cell.Barcode', 'Type', 'RNA_snn_res.0.1', 'RNA_snn_res.0.2', 'RNA_snn_res.0.3', 'RNA_snn_res.0.4', 'RNA_snn_res.0.5', 'RNA_snn_res.0.6', 'RNA_snn_res.0.7', 'RNA_snn_res.0.8', 'RNA_snn_res.0.9', 'RNA_snn_res.1', 'RNA_snn_res.1.1', 'RNA_snn_res.1.2', 'seurat_clusters', 'celltype', 'seurat_annotation'
# var: 'vf_vst_counts_mean', 'vf_vst_counts_variance', 'vf_vst_counts_variance.expected', 'vf_vst_counts_variance.standardized', 'vf_vst_counts_variable', 'vf_vst_counts_rank', 'var.features', 'var.features.rank'
# obsm: 'X_harmony', 'X_pca', 'X_umap'
ha5d格式数据转化成seruat对象
1.导入
rm(list = ls())
library(sceasy)
library(reticulate)
library(Seurat)
library(BiocParallel)
register(MulticoreParam(workers = 4, progressbar = TRUE))
2、R语言转换
# h5ad转为Seurat
sceasy::convertFormat(obj = "scRNA.h5ad", from="anndata",to="seurat",outFile = 'scRNA.rds')
# X -> counts
# An object of class Seurat
# 28269 features across 44651 samples within 1 assay
# Active assay: RNA (28269 features, 0 variable features)
# 2 layers present: counts, data
# 3 dimensional reductions calculated: pca, tsne, umap
这种方法得到的数据是SeruatV4版本的,所以如果要用于SeruatV5的话还需要再转化一下。
还有细胞数很多的话sceasy就不好用了,这个时候可以用dior包。
参考资料:
-
sceasy: https://github.com/cellgeni/sceasy
-
dior: https://github.com/JiekaiLab/dior
-
单细胞天地: https://mp.weixin.qq.com/s/qHBeQnYJdK0ATGlTOROPeA
-
生信菜鸟团: https://mp.weixin.qq.com/s/8fwJSc9Dnp8h_Suv76oXVA
-
KS科研分享与服务:https://mp.weixin.qq.com/s/Wt9TU5Qk3yqPDlRlXr6BfQ
注:若对内容有疑惑或者有发现明确错误的朋友,请联系后台(欢迎交流)。更多内容可关注公众号:生信方舟
- END -
相关文章:
方法学习和整理)
单细胞Seruat和h5ad数据格式互换(R与python)方法学习和整理
SeruatV4数据转化为h5ad格式数据 1、导入(R) rm(list ls()) library(Seurat) library(qs) library(reticulate) library(hdf5r) library(sceasy) library(BiocParallel) register(MulticoreParam(workers 4, progressbar TRUE)) scRNA <- qread("sc_dataset.qs&q…...

分布式难题-三座大山NPC
文章目录 1. 三座大山 NPC 的概念2. NPC 细分理解2.1. Network Delay 网络延迟2.2. Process Pause 进程暂停2.3. Clock Drift 时钟漂移Is the Algorithm Asynchronous? 本文参考: RedLock红锁安全性争论(上) https://martin.kleppmann.com/…...

两个方法教你设置Excel密码,防止修改和复制Excel表格内容
EXCEL是一款功能强大的电子表格软件,广泛用于各个地方。然而,对于一些重要的表格文件需要通过设置密码来限制大就的修改和复制权限。因而,对于一个EXCEL表格,通过密码设置大家有访问表格的权限,但无法修改数据的权限。…...

Java解析Excel文件
目录 背景 技术选型 开源Java框架选型 1. Apache POI 2. EasyExcel 收费Java框架选型 1. Spire.XLS for java 2. Aspose 总结 背景 在低代码产品的研发过程中,为用户提供数据导入导出的能力时,无法避免的就是对EXCEL解析的能力,所以本篇通过介…...

Require:基于雪花算法完成一个局部随机,全局离散没有热点切唯一的数值Id生成器。
【雪花算法】雪花算法(Snowflake Algorithm)是Twitter开源的用于生成唯一ID的算法,它可以在分布式系统中生成唯一的64位长整数ID。这种ID生成方式既保证了趋势递增,又保证了在不同数据中心、不同机器上生成的ID的唯一性。 符号位&…...

libevent - Macro function
TAILQ_INIT /** Tail queue functions.* 尾队列的头结点初始化为空队列。*/ #define TAILQ_INIT(head) do { \(head)->tqh_first NULL; \(head)->tqh_last &(head)->tqh_first; \ } while (/*CONSTCOND*/0)TAILQ_INIT 宏是一个用于初始化尾队列头部…...

408算法题leetcode--第17天
101. 对称二叉树 101. 对称二叉树思路:递归,对称即两个子树的左边和右边分别一样;一个子树是左中右遍历,另一个是右中左遍历;写的时候可以分三步,确定函数参数以及返回类型,确定终止条件&#…...

机器人顶刊IEEE T-RO发布无人机动态环境高效表征成果:基于粒子的动态环境连续占有地图
摘要:本研究有效提高了动态环境中障碍物建模的精度和效率。NOKOV度量动作捕捉系统助力评估动态占用地图在速度估计方面的性能。 近日,上海交通大学、荷兰代尔夫特理工研究团队在机器人顶刊IEEE T-RO上发表题为Continuous Occupancy Mapping in Dynamic …...

spring-boot web + vue
依赖的软件 maven 1. 官网下载zip 文件,比如apache-maven-3.9.9-bin.zip 2. 解压到某个盘符,必须保证父亲目录的名字包含英文,数字,破折号(-) 3. 设置环境变量M2_HOME, 并将%M2_HOME%\bin添加到windown…...

HDFS分布式文件系统01-HDFS架构与SHELL操作
HDFS分布式文件系统 学习目标第一课时知识点1-文件系统的分类单机文件系统网络文件系统分布式文件系统 知识点2-HDFS架构知识点3-HDFS的特点知识点4-HDFS的文件读写流程知识点5-HDFS的健壮性 第二课时知识点1-HDFS的Shell介绍HDFS Shell的语法格式如下。HDFS Shell客户端命令中…...

Go语言流程控制
Go语言流程控制 1.IF-ELSE2.Switch-Caseswitch 语句Type Switch 3.select 语句4.循环语句 1.IF-ELSE Go 编程语言中 if 语句的语法如下: if 布尔表达式 {/* 在布尔表达式为 true 时执行 */ }例如: package mainimport "fmt"func main() {va…...

无人机在救灾方面的应用!
一、灾害监测与评估 实时监测与评估:无人机可以快速到达灾害现场,通过搭载的高清摄像头、红外热成像仪等设备,对灾区进行实时监测和灾情评估。根据捕捉到的受灾范围、火势大小、建筑物损坏情况等关键信息,为救援行动提供决策依据…...

面试知识点总结篇一
一、C语言和C有什么区别 C语言是面向过程,强调用函数将问题分解为多个子任务,按顺序逐步进行。数据和操作分开C则是面向对象,面向对象是一种基于对象和类的编程范式,关注如何利用对象来抽象和模拟现实世界的实体。因此引入了类&a…...

【计算机网络 - 基础问题】每日 3 题(二十五)
✍个人博客:Pandaconda-CSDN博客 📣专栏地址:http://t.csdnimg.cn/fYaBd 📚专栏简介:在这个专栏中,我将会分享 C 面试中常见的面试题给大家~ ❤️如果有收获的话,欢迎点赞👍收藏&…...

【第十八章:Sentosa_DSML社区版-机器学习之协同过滤】
【第十八章:Sentosa_DSML社区版-机器学习之协同过滤】 1.算子介绍 协同过滤是推荐系统中常用的一种方法。该算法旨在填补用户-产品关联矩阵中缺少的项。在算法中,用户和产品都是通过一组少量的潜在因素描述,这些潜在因素可以用于预测用户-产…...
TDOA方法求二维坐标的MATLAB代码演示与讲解
引言 时间差定位(Time Difference of Arrival, TDOA)是一种用于确定信号源位置的技术,广泛应用于无线通信、声学定位等领域。通过测量信号到达多个接收器的时间差,可以计算出信号源的二维坐标。本文将通过MATLAB代码演示如何使用TDOA方法来求解二维坐标。 TDOA原理 TDOA…...

基于微信的原创音乐小程序的设计与实现+ssm论文源码调试讲解
第二章 开发工具及关键技术介绍 2.1 JAVA技术 Java主要采用CORBA技术和安全模型,可以在互联网应用的数据保护。它还提供了对EJB(Enterrise JavaBeans)的全面支持,java servlet AI,JS(java server ages&…...

基于大数据技术的颈椎病预防交流与数据分析及可视化系统
作者:计算机学姐 开发技术:SpringBoot、SSM、Vue、MySQL、JSP、ElementUI、Python、小程序等,“文末源码”。 专栏推荐:前后端分离项目源码、SpringBoot项目源码、Vue项目源码、SSM项目源码 精品专栏:Java精选实战项目…...

Spring MVC中实现一个文件上传和下载功能
说到文件上传和下载,相信每个开发者都有或多或少的接触过文件上传的功能吧,文件上传和下载是我们在学习计算机网络应用常见的一个功能,主要涉及到用户和服务器之间的数据传输。 我们来对文件上传和下载功能的进行相关概述吧! 文…...
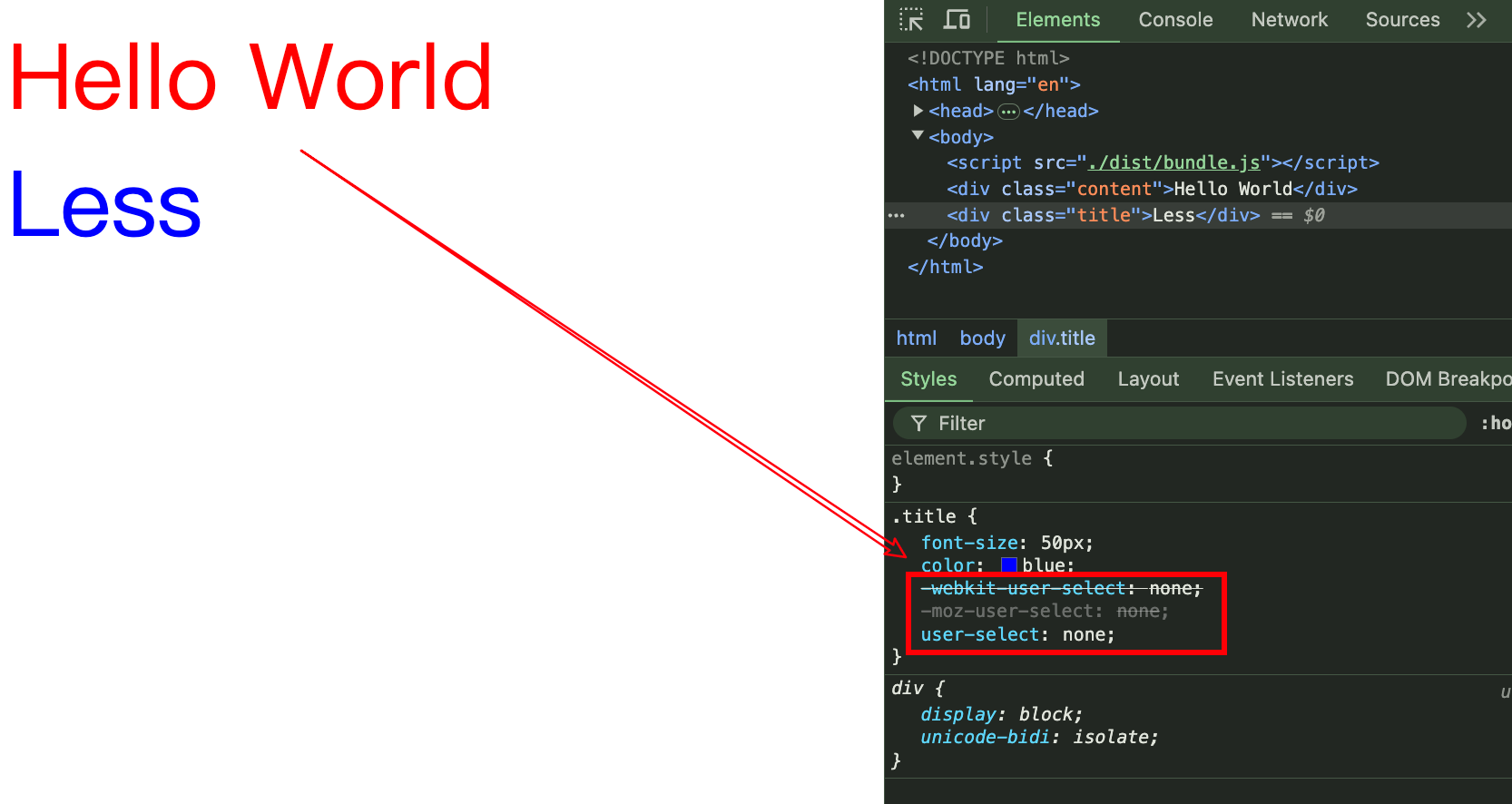
Webpack 介绍
Webpack 介绍 Date: August 29, 2024 全文概要 Webpack概念: Webpack是一个静态的模块化的打包工具,可以为现代的 JavaSript 应用程序进行打包。 1-静态:Webpack可以将代码打包成最终的静态资源 2-模块化:webpack支持各种模块…...
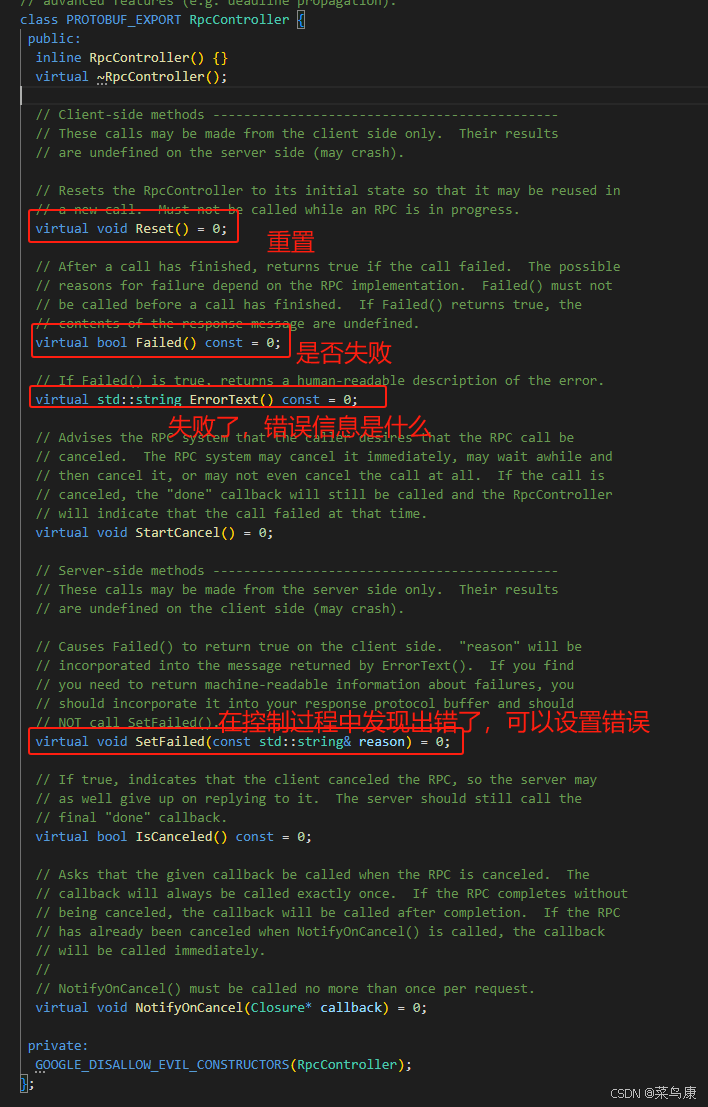
C++实现分布式网络通信框架RPC(3)--rpc调用端
目录 一、前言 二、UserServiceRpc_Stub 三、 CallMethod方法的重写 头文件 实现 四、rpc调用端的调用 实现 五、 google::protobuf::RpcController *controller 头文件 实现 六、总结 一、前言 在前边的文章中,我们已经大致实现了rpc服务端的各项功能代…...

【Linux】shell脚本忽略错误继续执行
在 shell 脚本中,可以使用 set -e 命令来设置脚本在遇到错误时退出执行。如果你希望脚本忽略错误并继续执行,可以在脚本开头添加 set e 命令来取消该设置。 举例1 #!/bin/bash# 取消 set -e 的设置 set e# 执行命令,并忽略错误 rm somefile…...
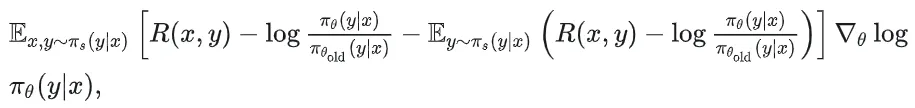
51c自动驾驶~合集58
我自己的原文哦~ https://blog.51cto.com/whaosoft/13967107 #CCA-Attention 全局池化局部保留,CCA-Attention为LLM长文本建模带来突破性进展 琶洲实验室、华南理工大学联合推出关键上下文感知注意力机制(CCA-Attention),…...

关于iview组件中使用 table , 绑定序号分页后序号从1开始的解决方案
问题描述:iview使用table 中type: "index",分页之后 ,索引还是从1开始,试过绑定后台返回数据的id, 这种方法可行,就是后台返回数据的每个页面id都不完全是按照从1开始的升序,因此百度了下,找到了…...

【Web 进阶篇】优雅的接口设计:统一响应、全局异常处理与参数校验
系列回顾: 在上一篇中,我们成功地为应用集成了数据库,并使用 Spring Data JPA 实现了基本的 CRUD API。我们的应用现在能“记忆”数据了!但是,如果你仔细审视那些 API,会发现它们还很“粗糙”:有…...

JDK 17 新特性
#JDK 17 新特性 /**************** 文本块 *****************/ python/scala中早就支持,不稀奇 String json “”" { “name”: “Java”, “version”: 17 } “”"; /**************** Switch 语句 -> 表达式 *****************/ 挺好的ÿ…...
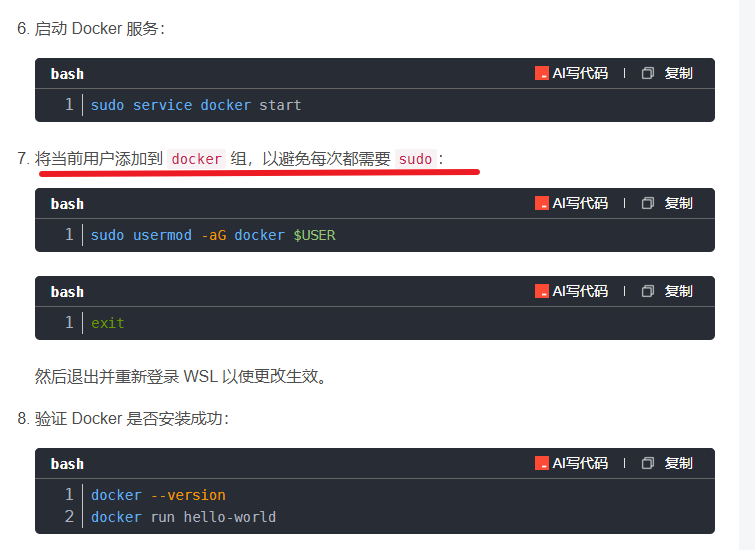
在WSL2的Ubuntu镜像中安装Docker
Docker官网链接: https://docs.docker.com/engine/install/ubuntu/ 1、运行以下命令卸载所有冲突的软件包: for pkg in docker.io docker-doc docker-compose docker-compose-v2 podman-docker containerd runc; do sudo apt-get remove $pkg; done2、设置Docker…...

tree 树组件大数据卡顿问题优化
问题背景 项目中有用到树组件用来做文件目录,但是由于这个树组件的节点越来越多,导致页面在滚动这个树组件的时候浏览器就很容易卡死。这种问题基本上都是因为dom节点太多,导致的浏览器卡顿,这里很明显就需要用到虚拟列表的技术&…...

CMake控制VS2022项目文件分组
我们可以通过 CMake 控制源文件的组织结构,使它们在 VS 解决方案资源管理器中以“组”(Filter)的形式进行分类展示。 🎯 目标 通过 CMake 脚本将 .cpp、.h 等源文件分组显示在 Visual Studio 2022 的解决方案资源管理器中。 ✅ 支持的方法汇总(共4种) 方法描述是否推荐…...
Reasoning over Uncertain Text by Generative Large Language Models
https://ojs.aaai.org/index.php/AAAI/article/view/34674/36829https://ojs.aaai.org/index.php/AAAI/article/view/34674/36829 1. 概述 文本中的不确定性在许多语境中传达,从日常对话到特定领域的文档(例如医学文档)(Heritage 2013;Landmark、Gulbrandsen 和 Svenevei…...
