KofamScan-KEGG官方推荐的使用系同源和隐马尔可夫模型进行KO注释
文章目录
- 简介
- 安装
- 使用
- 输入蛋白序列
- 输出detail-tsv格式
- 输出detail格式
- 输出mapper格式
- 输出结果
- detail和detail-tsv格式
- mapper格式
- 常用命令
- tmp目录
- 与emapper结果比较
- 其他参数
- 参考
简介
KofamScan 是一款基于 KEGG 直系同源和隐马尔可夫模型(HMM)的基因功能注释工具,可通过同源搜索和预先计算的自适应分数阈值,将 KEGG 同源物(KOs)分配给蛋白质序列的隐马尔可夫模型(KOfam)数据库。在线版本可在 https://www.genome.jp/tools/kofamkoala/ 上获取。KofamKOALA 比现有的 KO 分配工具更快,其准确性可与性能最好的工具相媲美。KofamKOALA 的功能注释有助于将基因与 KEGG 通路图等 KEGG 资源联系起来,并促进分子网络的重建。
安装
# 软件
(base) [yut@io02 ~]$ mamba install -c bioconda kofamscan
# 数据库
# filezila打开ftp.genome.jp看一下最新版数据库更新时间
mkdir -p KoFamDB20231025
cd KoFamDB20231025
#数据库:
wget ftp://ftp.genome.jp/pub/db/kofam/ko_list.gz
wget ftp://ftp.genome.jp/pub/db/kofam/profiles.tar.gz
gunzip ko_list.gz
tar xvzf profiles.tar.gz
使用
输入蛋白序列
查询文件是包含一个或多个氨基酸序列的 FASTA 文件。不能使用核苷酸序列。每个序列必须有一个唯一的名称。序列名称是介于标题符号(“>”)和第一个空白字符(空格、制表符、换行符等)之间的字符串。请勿在">"之后使用空格。
(base) [yut@node01 TestData]$ grep -c ">" GCA_002343985.1_ASM234398v1_genomic.faa
2486 # 蛋白数量
输出detail-tsv格式
time exec_annotation -p ~/Database/KoFamDB20231025/profiles -k ~/Database/KoFamDB20231025/ko_list --cpu 10 --tmp-dir . -E 1e-5 -f detail-tsv -o GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail-tsv.tsv GCA_002343985.1_ASM234398v1_genomic.faa &>GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail.log# -p与-k为上述数据库配置文件
# -E 为e-value
# -f为输出格式,包括:
-f, --format <format> Format of the output [detail]detail: Detail for each hits (including hits below threshold)detail-tsv: Tab separeted values for detail formatmapper: KEGG Mapper compatible formatmapper-one-line: Similar to mapper, but all hit KOs are listed in one line
# 3m18.871s
输出detail格式
(base) [yut@node01 TestData]$ time exec_annotation -p ~/Database/KoFamDB20231025/profiles -k ~/Database/KoFamDB20231025/ko_list --cpu 10 --tmp-dir . -E 1e-5 -f detail -o GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail_format.tsv GCA_002343985.1_ASM234398v1_genomic.faa &>GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail.log&
# real 3m0.691s
输出mapper格式
(base) [yut@node01 TestData]$ time exec_annotation -p ~/Database/KoFamDB20231025/profiles -k ~/Database/KoFamDB20231025/ko_list --cpu 10 --tmp-dir . -E 1e-5 -f mapper -o GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_format.tsv GCA_002343985.1_ASM234398v1_genomic.faa &>GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper.log&
# real 3m13.254s
输出结果
detail和detail-tsv格式
(base) [yut@node01 TestData]$ head GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail-tsv.tsv
# gene name KO thrshld score E-value "KO definition"
# --------- ------ ------- ------ --------- -------------
* GLGNCOCG_00001 K09946 28.63 105.8 3.2e-31 "uncharacterized protein"
* GLGNCOCG_00002 K22441 90.53 175.7 2.4e-52 "diamine N-acetyltransferase [EC:2.3.1.57]"GLGNCOCG_00002 K03789 105.10 66.4 4.4e-19 "[ribosomal protein S18]-alanine N-acetyltransferase [EC:2.3.1.266]"GLGNCOCG_00002 K03828 117.57 66.1 4.1e-19 "putative acetyltransferase [EC:2.3.1.-]"GLGNCOCG_00002 K03825 113.37 57.6 1.7e-16 "L-phenylalanine/L-methionine N-acetyltransferase [EC:2.3.1.53 2.3.1.-]"GLGNCOCG_00002 K24217 175.13 55.7 6e-16 "L-amino acid N-acyltransferase [EC:2.3.1.-]"GLGNCOCG_00002 K09994 117.73 50.8 2.6e-14 "(aminoalkyl)phosphonate N-acetyltransferase [EC:2.3.1.280]"GLGNCOCG_00002 K15520 93.30 50.6 3.5e-14 "mycothiol synthase [EC:2.3.1.189]"
(base) [yut@node01 TestData]$ head GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail_format.tsv
# gene name KO thrshld score E-value KO definition
#-------------------- ------ ------- ------ --------- ---------------------
* GLGNCOCG_00001 K09946 28.63 105.8 3.2e-31 uncharacterized protein
* GLGNCOCG_00002 K22441 90.53 175.7 2.4e-52 diamine N-acetyltransferase [EC:2.3.1.57]GLGNCOCG_00002 K03789 105.10 66.4 4.4e-19 [ribosomal protein S18]-alanine N-acetyltransferase [EC:2.3.1.266]GLGNCOCG_00002 K03828 117.57 66.1 4.1e-19 putative acetyltransferase [EC:2.3.1.-]GLGNCOCG_00002 K03825 113.37 57.6 1.7e-16 L-phenylalanine/L-methionine N-acetyltransferase [EC:2.3.1.53 2.3.1.-]GLGNCOCG_00002 K24217 175.13 55.7 6e-16 L-amino acid N-acyltransferase [EC:2.3.1.-]GLGNCOCG_00002 K09994 117.73 50.8 2.6e-14 (aminoalkyl)phosphonate N-acetyltransferase [EC:2.3.1.280]GLGNCOCG_00002 K15520 93.30 50.6 3.5e-14 mycothiol synthase [EC:2.3.1.189](base) [yut@node01 TestData]$ sort -t $'\t' -k4 -n GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail-tsv.tsv|head
# --------- ------ ------- ------ --------- -------------
# gene name KO thrshld score E-value "KO definition"GLGNCOCG_01738 K19152 0.5 1.9e+02 "small toxic protein IbsD"GLGNCOCG_02049 K21664 1.7 4.3 "KSHV latency-associated nuclear antigen"GLGNCOCG_02331 K21664 2.9 1.8 "KSHV latency-associated nuclear antigen"GLGNCOCG_00788 K21664 4.7 0.53 "KSHV latency-associated nuclear antigen"GLGNCOCG_01757 K19313 5.6 1.5 "aminocyclitol acetyltransferase [EC:2.3.-.-]"GLGNCOCG_01062 K16402 6.8 0.023 "soraphen polyketide synthase B"GLGNCOCG_00067 K16007 7.1 0.033 "8,8a-deoxyoleandolide synthase 1"GLGNCOCG_01945 K26748 7.2 0.27 "MFS transporter, AAHS family, 2,4-dichlorophenoxyacetate transporter"# 有明确KO号的基因数量
(base) [yut@node01 TestData]$ awk '$1~/*/' GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail-tsv.tsv|wc -l
1216(base) [yut@node01 TestData]$ echo|awk '{print 1216/2486*100}'
48.9139
可以看到detailj及detail-tsv格式是用制表符将所有比对的结果都输出,即除了满足阈值< 1e-5,同时也将大于该阈值的结果输出。总共7列,第一列是标记该基因是否分配到Knumber,带星号的表示有Knumber;第二列表示输入基因名称;第三列是KO号;第四列是阈值;第五列是得分,得分一般大于前面阈值列的就会有KO;第六列是E-value;第七列是KO definition。该基因组最终有48.91%的蛋白序列有KO注释。
mapper格式
(base) [yut@node01 TestData]$ head GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_format.tsv
GLGNCOCG_00001 K09946
GLGNCOCG_00002 K22441
GLGNCOCG_00003
GLGNCOCG_00004 K03286
GLGNCOCG_00005
GLGNCOCG_00006
GLGNCOCG_00007
GLGNCOCG_00008
GLGNCOCG_00009
GLGNCOCG_00010# 一个基因注释到多个KO
(base) [yut@node01 TestData]$ grep GLGNCOCG_00297 GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_format.tsv
GLGNCOCG_00297 K13990
GLGNCOCG_00297 K00603
GLGNCOCG_00297 K01500
(base) [yut@node01 TestData]$ grep GLGNCOCG_00297 GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_detail_format.tsv
* GLGNCOCG_00297 K13990 507.67 749.9 4.5e-226 glutamate formiminotransferase / formiminotetrahydrofolate cyclodeaminase [EC:2.1.2.5 4.3.1.4]
* GLGNCOCG_00297 K00603 405.93 411.8 4.9e-124 glutamate formiminotransferase / 5-formyltetrahydrofolate cyclo-ligase [EC:2.1.2.5 6.3.3.2]
* GLGNCOCG_00297 K01500 201.63 215.6 7.4e-65 methenyltetrahydrofolate cyclohydrolase [EC:3.5.4.9]GLGNCOCG_00297 K01746 429.53 65.3 8.3e-19 formiminotetrahydrofolate cyclodeaminase [EC:4.3.1.4]GLGNCOCG_00297 K10740 25.77 10.7 0.042 replication factor A3# 有KO的
(base) [yut@node01 TestData]$ awk -F"\t" '$2!=""' GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_format.tsv|wc -l
1216
mapper格式总共两列,第一列是基因ID,第二列是Knumber,没有注释到的为空,注意一个基因可能同时注释到多个KO。
- mapper-one-line格式:类似上面的mapper格式,不一样的是一个基因如果有多个KO则以制表符分割放在同一行。
(base) [yut@node01 TestData]$ less GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper-one-line_format.tsv
GLGNCOCG_00294 K07304
GLGNCOCG_00295 K01924
GLGNCOCG_00296 K01468
GLGNCOCG_00297 K13990 K00603 K01500
GLGNCOCG_00298
GLGNCOCG_00299
GLGNCOCG_00300
GLGNCOCG_00301
GLGNCOCG_00302
(base) [yut@node01 TestData]$ awk 'NF>2' GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper-one-line_format.tsv|cat -A
GLGNCOCG_00069^IK13693^IK01079$
GLGNCOCG_00108^IK01417^IK20276$
GLGNCOCG_00297^IK13990^IK00603^IK01500$
GLGNCOCG_00486^IK03668^IK06079$
GLGNCOCG_00489^IK06079^IK03668$
GLGNCOCG_00776^IK00027^IK00029$
GLGNCOCG_00998^IK25153^IK25151$
常用命令
参数:evalue:1e-5(the ORFs were searched against the Kyoto Encyclopedia of Genes and Genomes (KEGG) database [48] using KofamScan version 1.2.0 (E-value < 10−5, score>predefined thresholds by KofamScan) [49]. 参考https://www.nature.com/articles/s41396-023-01546-2,https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-023-01621-y)
每行一个基因,一个基因多个KO则多行输出,不输出注释不到的基因,
(base) [yut@node01 TestData]$ time exec_annotation -p ~/Database/KoFamDB20231025/profiles -k ~/Database/KoFamDB20231025/ko_list --cpu 10 -E 1e-5 -f mapper --no-report-unannotated -o GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_annot_format_1e- 5.tsv GCA_002343985.1_ASM234398v1_genomic.faa &
tmp目录
- 不写 --tmp-dir则默认在当前目录创建tmp目录,其下生成tabular目录,里面是所有KO的HMM信息。
- 注意:如果使用
parallel同时并行多个文件时,一定要为每个指定一个不同名称的目录,否则则会互相影响,输出不完整。
(base) [yut@node01 TestData]$ ls tmp
tabular
(base) [yut@node01 TestData]$ ll tmp/tabular/|head
总用量 51M
-rw-r--r-- 1 yut lzdx 2.4K 11月 16 11:17 K00001
-rw-r--r-- 1 yut lzdx 1.2K 11月 16 11:17 K00002
-rw-r--r-- 1 yut lzdx 2.3K 11月 16 11:17 K00003
-rw-r--r-- 1 yut lzdx 2.9K 11月 16 11:17 K00004
-rw-r--r-- 1 yut lzdx 1.4K 11月 16 11:17 K00005
-rw-r--r-- 1 yut lzdx 1.2K 11月 16 11:17 K00006
-rw-r--r-- 1 yut lzdx 997 11月 16 11:17 K00007
-rw-r--r-- 1 yut lzdx 2.8K 11月 16 11:17 K00008
-rw-r--r-- 1 yut lzdx 1.2K 11月 16 11:17 K00009
(base) [yut@node01 TestData]$ head tmp/tabular/K00001
# --- full sequence ---- --- best 1 domain ---- --- domain number estimation ----
# target name accession query name accession E-value score bias E-value score bias exp reg clu ov env dom rep inc description of target
#------------------- ---------- -------------------- ---------- --------- ------ ----- --------- ------ ----- --- --- --- --- --- --- --- --- ---------------------
GLGNCOCG_00164 - K00001 - 1.3e-52 177.0 0.4 1.5e-52 176.8 0.4 1.0 1 0 0 1 1 1 1 NADP-dependent alcohol dehydrogenase C
GLGNCOCG_00550 - K00001 - 8.8e-48 161.1 1.0 9.8e-48 160.9 1.0 1.0 1 0 0 1 1 1 1 Alcohol dehydrogenase
GLGNCOCG_02093 - K00001 - 1.1e-05 22.2 3.7 1.3e-05 21.9 3.7 1.1 1 0 0 1 1 1 1 NAD(P) transhydrogenase subunit alpha part 1
GLGNCOCG_00799 - K00001 - 0.00063 16.4 1.5 0.16 8.4 0.3 2.2 2 0 0 2 2 2 2 Glutamate synthase [NADPH] small chain
GLGNCOCG_01761 - K00001 - 0.0011 15.6 0.9 0.0019 14.8 0.3 1.6 2 0 0 2 2 2 1 Alanine dehydrogenase 2
GLGNCOCG_01061 - K00001 - 0.0035 13.9 0.0 0.0057 13.2 0.0 1.4 1 0 0 1 1 1 1 UDP-N-acetyl-D-glucosamine 6-dehydrogenase
GLGNCOCG_01051 - K00001 - 0.0053 13.3 0.3 0.0077 12.8 0.3 1.2 1 0 0 1 1 1 1 Glutamate dehydrogenase与emapper结果比较
## emapper-2.1.9,evalue:default, 0.001;eggnog_5.02_database/eggnog_proteins.dmnd
## /apps/anaconda3/envs/eggnog-mapper-2.1.9/bin/emapper.py -i GCA_002343985.1_ASM234398v1_genomic.faa --cpu 20 -o emapper_out --override --temp_dir .
(base) [yut@node01 TestData]$ awk -F"\t" 'NR>1{print $1,$12}' emapper_out.emapper.annotations|grep ko|grep -v "#"|wc -l
1232## kofamscan: evalue 1e-3
(base) [yut@node01 TestData]$ time exec_annotation -p ~/Database/KoFamDB20231025/profiles -k ~/Database/KoFamDB20231025/ko_list --cpu 10 -E 1e-3 -f mapper --no-report-unannotated -o GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_annot_format_1e-3.tsv GCA_002343985.1_ASM234398v1_genomic.faa &(base) [yut@node01 TestData]$ wc -l GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_annot_format_1e-3.tsv
1218 GCA_002343985.1_ASM234398v1_genomic.faa_kofamscan_mapper_annot_format_1e-3.tsv
emapper在1e-3的阈值下要比kofamscan在1e-5多16个基因的KO注释,然而当降低kofamscan的evalue到1e-3也仅多了两个基因的KO。
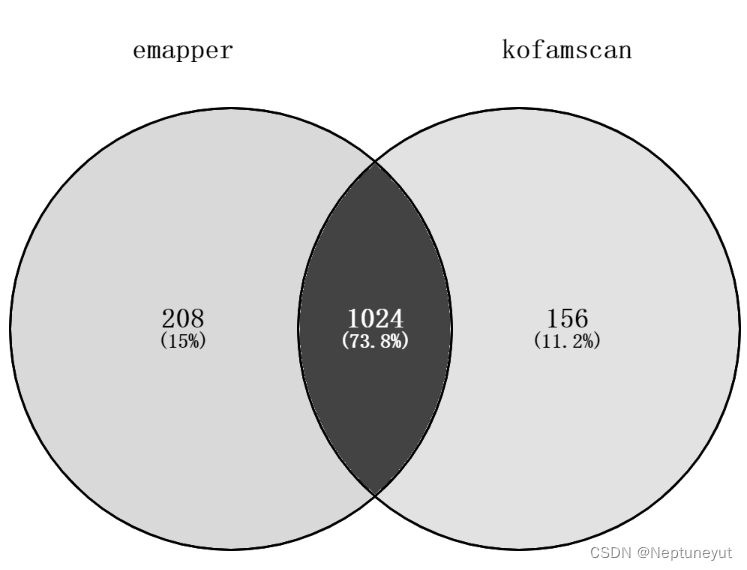
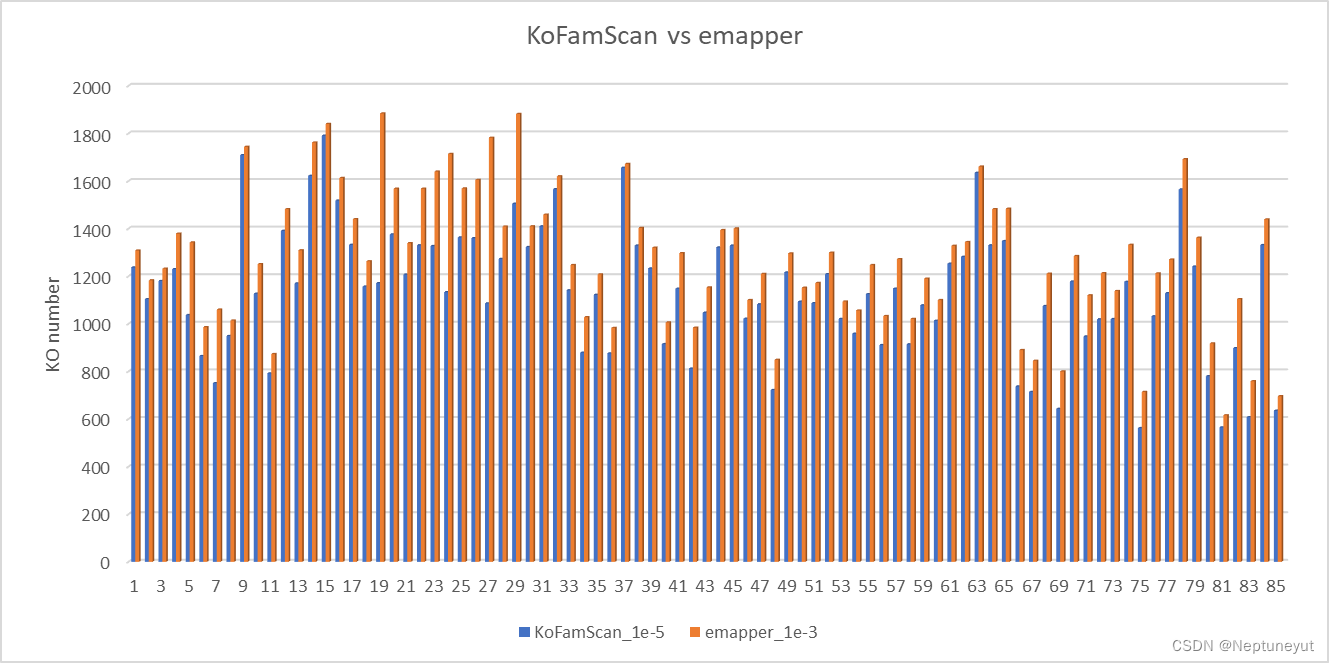
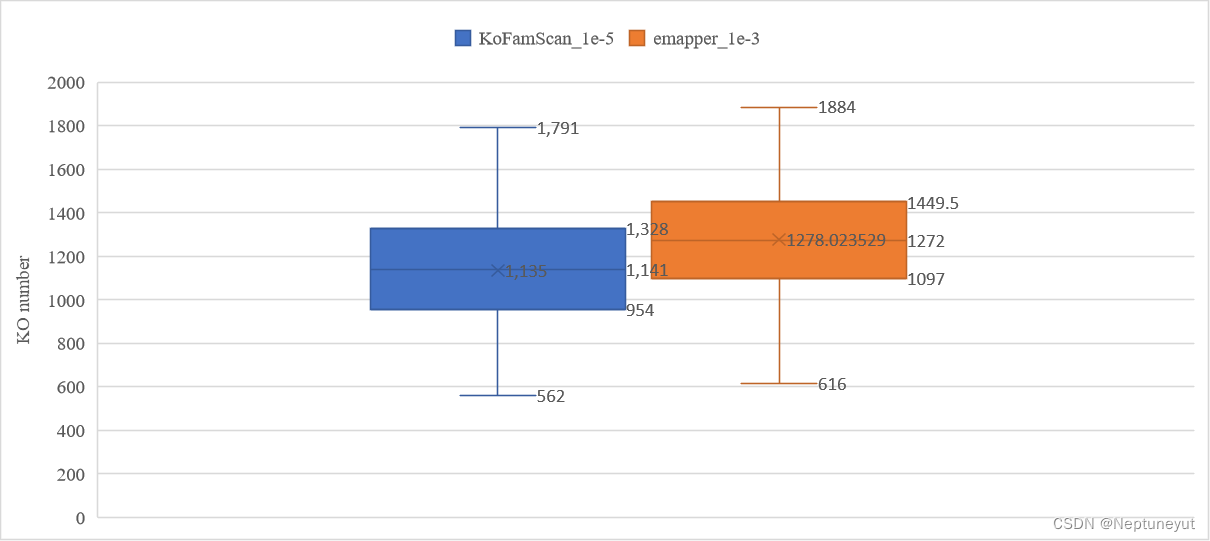
- 对另外多个细菌基因组注释结果统计
其他参数
Usage: exec_annotation [options] <query><query> FASTA formatted query sequence file-o <file> File to output the result [stdout]-p, --profile <path> Profile HMM database-k, --ko-list <file> KO information file--cpu <num> Number of CPU to use [1]-c, --config <file> Config file--tmp-dir <dir> Temporary directory [./tmp] # 不写的话默认是当前目录下创建tmp-E, --e-value <e_value> Largest E-value required of the hits-T, --threshold-scale <scale>The score thresholds will be multiplied by this value-f, --format <format> Format of the output [detail]detail: Detail for each hits (including hits below threshold) # 按照hit展示,一个基因可能有多个hits,默认格式。显示基因名称、分配的 K 编号、KO 的阈值、hmmsearch 分数和 E 值以及 KO 的定义。此外,如果得分高于阈值,则在行首添加星号 "*"。detail-tsv: Tab separeted values for detail format # 制表符分隔与上面一样mapper: KEGG Mapper compatible format # 两列,基因与KOmapper-one-line: Similar to mapper, but all hit KOs are listed in one line # 一个基因的多个hits在一行--[no-]report-unannotated Sequence name will be shown even if no KOs are assignedDefault is true when format=mapper or mapper-all,false when format=detail--create-alignment Create domain annotation files for each sequenceThey will be located in the tmp directoryIncompatible with -r-r, --reannotate Skip hmmsearchIncompatible with --create-alignment--keep-tabular Neither create tabular.txt nor delete K number filesBy default, all K number files will be combined intoa tabular.txt and delete them--keep-output Neither create output.txt nor delete K number filesBy default, all K number files will be combined intoa output.txt and delete themMust be with --create-alignment-h, --help Show this message and exit参考
Kofamscan
KofamKOALA: KEGG Ortholog assignment based on profile HMM and adaptive score threshold
IF: 5.8 Q1 B3
相关文章:

KofamScan-KEGG官方推荐的使用系同源和隐马尔可夫模型进行KO注释
文章目录 简介安装使用输入蛋白序列输出detail-tsv格式输出detail格式输出mapper格式 输出结果detail和detail-tsv格式mapper格式常用命令tmp目录 与emapper结果比较其他参数参考 简介 KofamScan 是一款基于 KEGG 直系同源和隐马尔可夫模型(HMM)的基因功…...
代码随想录算法训练营第五十五天丨 动态规划part16
583. 两个字符串的删除操作 思路 #动态规划一 本题和动态规划:115.不同的子序列 (opens new window)相比,其实就是两个字符串都可以删除了,情况虽说复杂一些,但整体思路是不变的。 这次是两个字符串可以相互删了,这…...

【Linux】kernel与应用消息队列的一种设计
Linux进程间通讯的方式有很多种,这里介绍一种通过消息队列的方式来实现kernel与APP之间的消息收发实现方式,这种方式特别适用于,kernel中发送消息,应用层收取消息。 消息队列设备驱动 该方法的设计思路即是创建一个消息队列的设…...

我们常说的网络资产,具体是如何定义的?
文章目录 什么叫网络资产?官方定义的网络资产网络资产数字化定义推荐阅读 什么叫网络资产? 通过百度查询搜索什么叫网络资产?大体上都将网络资产归类为计算机网络中的各类设备。 基本上会定义网络传输通信架构中用到的主机、网络设备、防火…...

WPF中可冻结对象
在WPF(Windows Presentation Foundation)中,"可冻结对象"指的是那些在创建之后可以被设置为不可更改状态的对象。这种特性允许这些对象更有效地被共享和复制,并且可以增加性能。 例如,Brushes,P…...
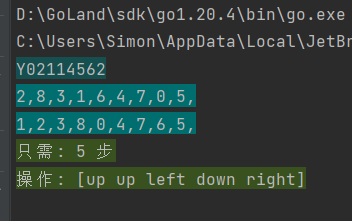
【人工智能实验】A*算法求解8数码问题 golang
人工智能经典问题八数码求解 实际上是将求解转为寻找最优节点的问题,算法流程如下: 求非0元素的逆序数的和,判断是否有解将开始状态放到节点集,并设置访问标识位为true从节点集中取出h(x)g(x)最小的节点判断取出的节点的状态是不…...
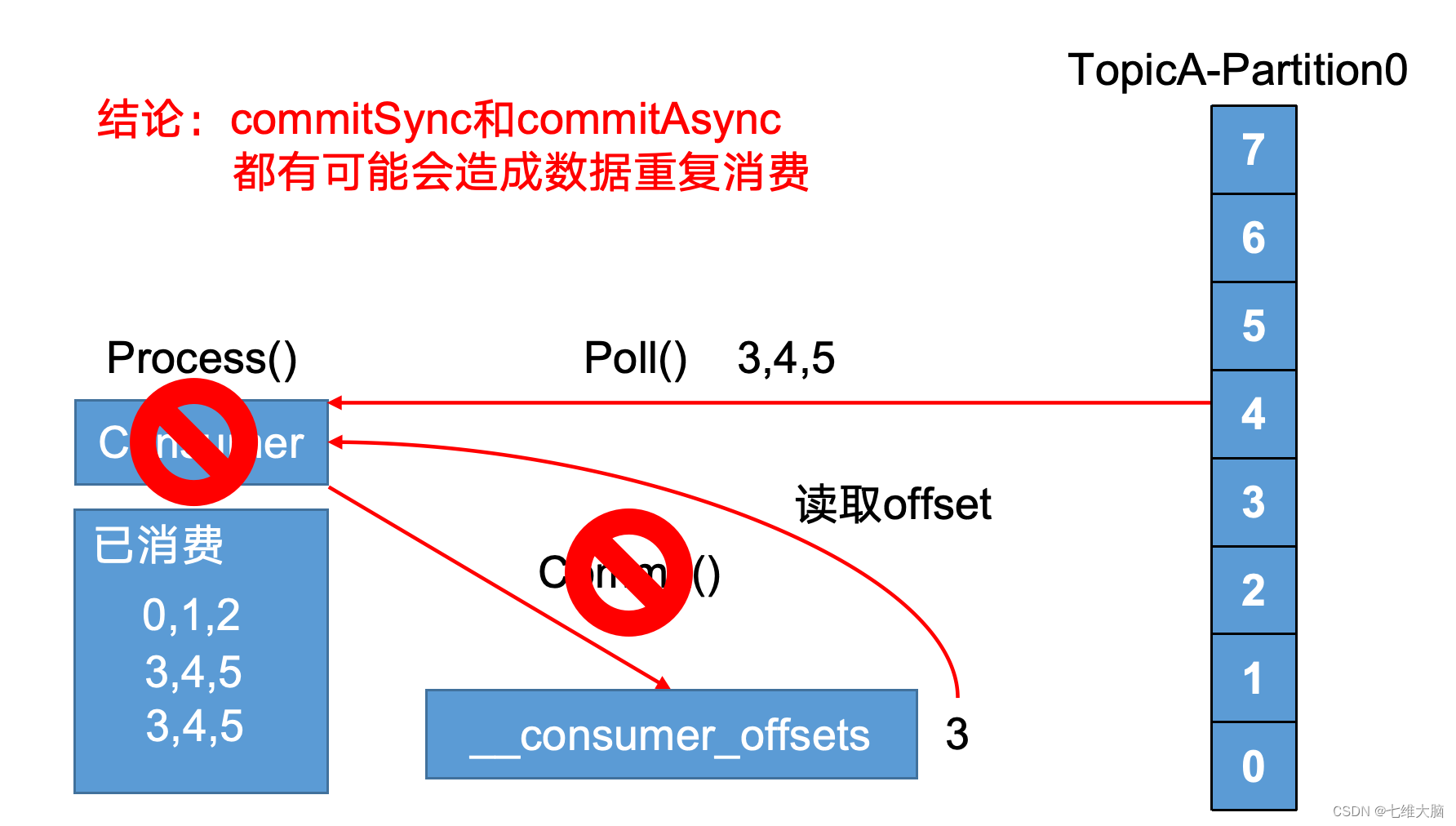
Kafka学习笔记(二)
目录 第3章 Kafka架构深入3.3 Kafka消费者3.3.1 消费方式3.3.2 分区分配策略3.3.3 offset的维护 3.4 Kafka高效读写数据3.5 Zookeeper在Kafka中的作用3.6 Kafka事务3.6.1 Producer事务3.6.2 Consumer事务(精准一次性消费) 第4章 Kafka API4.1 Producer A…...
Typora for Mac:打造全新文本编辑体验
Typora for Mac是一款与众不同的文本编辑器,它不仅拥有直观易用的界面,还融合了Markdown语法和富文本编辑的功能,为用户带来了前所未有的写作和编辑体验。 一、简洁明了的界面设计 Typora for Mac的界面简洁明了,让用户可以专注…...

TikTok与媒体素养:如何辨别虚假信息?
在当今数字时代,社交媒体平台如TikTok已经成为信息传播和社交互动的主要渠道之一。然而,随之而来的是虚假信息的泛滥,这对用户的媒体素养提出了严峻的挑战。本文将探讨TikTok平台上虚假信息的现象,以及如何提高媒体素养࿰…...
Spring Boot 中使用 ResourceLoader 加载资源的完整示例
ResourceLoader 是 Spring 框架中用于加载资源的接口。它定义了一系列用于获取资源的方法,可以处理各种资源,包括类路径资源、文件系统资源、URL 资源等。 以下是 ResourceLoader 接口的主要方法: Resource getResource(String location)&am…...
1688往微信小程序自营商城铺货商品采集API接口
一、背景介绍 随着移动互联网的快速发展,微信小程序作为一种新型的电商形态,正逐渐成为广大商家拓展销售渠道、提升品牌影响力的重要平台。然而,对于许多传统企业而言,如何将商品信息快速、准确地铺货到微信小程序自营商城是一个…...

QStatusBar开发详解
一、QStatusBar接口说明 QStatusBar 类是 Qt 中用于创建和管理状态栏的类。它继承自 QFrame 类,提供了在主窗口底部显示消息、进度等信息的功能。以下是一些 QStatusBar 类的重要接口: 1.1 QStatusBar构造函数 QStatusBar(QWidget *parent nullptr);…...

后端接口性能优化分析-程序结构优化
👏作者简介:大家好,我是爱吃芝士的土豆倪,24届校招生Java选手,很高兴认识大家📕系列专栏:Spring源码、JUC源码🔥如果感觉博主的文章还不错的话,请👍三连支持&…...
【SpringBoot3+Vue3】三【实战篇】-后端(优化)
目录 一、登录优化-redis 1、SpringBoot集成redis 1.1 pom 1.2 yml 1.3 测试程序(非必须) 1.4 启动redis,执行测试程序 2、令牌主动失效(代码优化) 2.1 UserController设置token到redis 2.2 登录拦截器Log…...
DevExpress中文教程 - 如何在macOS和Linux (CTP)上创建、修改报表(上)
DevExpress Reporting是.NET Framework下功能完善的报表平台,它附带了易于使用的Visual Studio报表设计器和丰富的报表控件集,包括数据透视表、图表,因此您可以构建无与伦比、信息清晰的报表。 DevExpress Reports — 跨平台报表组件&#x…...
一个iOS tableView 滚动标题联动效果的实现
效果图 情景 tableview 是从屏幕顶部开始的,现在有导航栏,和栏目标题视图将tableView的顶部覆盖了 分析 我们为了达到滚动到某个分区选中标题的效果,就得知道 展示最顶部的cell或者区头在哪个分区范围内 所以我们必须首先获取顶部的位置 …...

代码执行相关函数以及简单例题
代码/命令 执行系列 相关函数 (代码注入)...
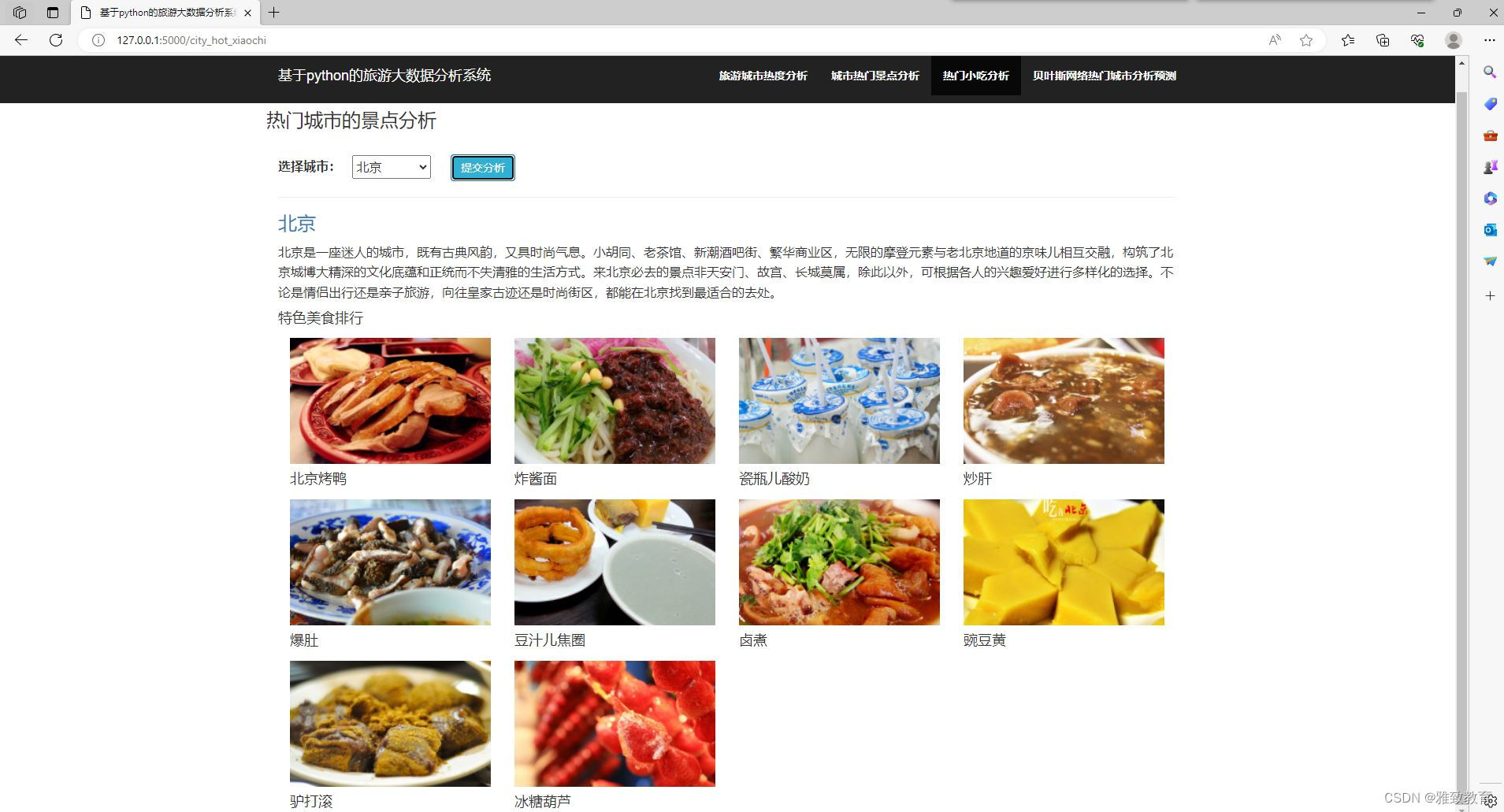
大数据爬虫分析基于Python+Django旅游大数据分析系统
欢迎大家点赞、收藏、关注、评论啦 ,由于篇幅有限,只展示了部分核心代码。 文章目录 一项目简介 二、功能三、系统四. 总结 一项目简介 基于Python和Django的旅游大数据分析系统是一种使用Python编程语言和Django框架开发的系统,用于处理和分…...

C# 结构体介绍
文章目录 定义结构体实例化结构体结构体的值类型特性结构体和类的区别限制 C# 中的结构体(Struct)是一种值类型数据结构,用于封装不同或相同类型的数据成一个单一的实体。结构体非常适合用来表示轻量级的对象,比如坐标点、颜色值或…...
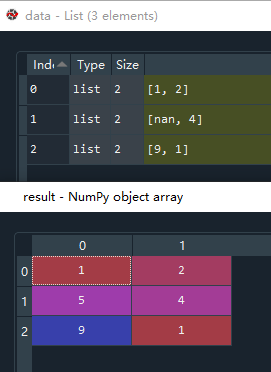
【机器学习】特征工程:特征预处理,归一化、标准化、处理缺失值
特征预处理采用的是特定的统计方法(数学方法)将数据转化为算法要求的数字 1. 数值型数据 归一化,将原始数据变换到[0,1]之间 标准化,数据转化到均值为0,方差为1的范围内 缺失值,缺失值处理成均值、中…...
渗透实战PortSwigger靶场-XSS Lab 14:大多数标签和属性被阻止
<script>标签被拦截 我们需要把全部可用的 tag 和 event 进行暴力破解 XSS cheat sheet: https://portswigger.net/web-security/cross-site-scripting/cheat-sheet 通过爆破发现body可以用 再把全部 events 放进去爆破 这些 event 全部可用 <body onres…...

条件运算符
C中的三目运算符(也称条件运算符,英文:ternary operator)是一种简洁的条件选择语句,语法如下: 条件表达式 ? 表达式1 : 表达式2• 如果“条件表达式”为true,则整个表达式的结果为“表达式1”…...
-----深度优先搜索(DFS)实现)
c++ 面试题(1)-----深度优先搜索(DFS)实现
操作系统:ubuntu22.04 IDE:Visual Studio Code 编程语言:C11 题目描述 地上有一个 m 行 n 列的方格,从坐标 [0,0] 起始。一个机器人可以从某一格移动到上下左右四个格子,但不能进入行坐标和列坐标的数位之和大于 k 的格子。 例…...

macOS多出来了:Google云端硬盘、YouTube、表格、幻灯片、Gmail、Google文档等应用
文章目录 问题现象问题原因解决办法 问题现象 macOS启动台(Launchpad)多出来了:Google云端硬盘、YouTube、表格、幻灯片、Gmail、Google文档等应用。 问题原因 很明显,都是Google家的办公全家桶。这些应用并不是通过独立安装的…...
MySQL 8.0 OCP 英文题库解析(十三)
Oracle 为庆祝 MySQL 30 周年,截止到 2025.07.31 之前。所有人均可以免费考取原价245美元的MySQL OCP 认证。 从今天开始,将英文题库免费公布出来,并进行解析,帮助大家在一个月之内轻松通过OCP认证。 本期公布试题111~120 试题1…...

【学习笔记】深入理解Java虚拟机学习笔记——第4章 虚拟机性能监控,故障处理工具
第2章 虚拟机性能监控,故障处理工具 4.1 概述 略 4.2 基础故障处理工具 4.2.1 jps:虚拟机进程状况工具 命令:jps [options] [hostid] 功能:本地虚拟机进程显示进程ID(与ps相同),可同时显示主类&#x…...

高效线程安全的单例模式:Python 中的懒加载与自定义初始化参数
高效线程安全的单例模式:Python 中的懒加载与自定义初始化参数 在软件开发中,单例模式(Singleton Pattern)是一种常见的设计模式,确保一个类仅有一个实例,并提供一个全局访问点。在多线程环境下,实现单例模式时需要注意线程安全问题,以防止多个线程同时创建实例,导致…...

Linux nano命令的基本使用
参考资料 GNU nanoを使いこなすnano基础 目录 一. 简介二. 文件打开2.1 普通方式打开文件2.2 只读方式打开文件 三. 文件查看3.1 打开文件时,显示行号3.2 翻页查看 四. 文件编辑4.1 Ctrl K 复制 和 Ctrl U 粘贴4.2 Alt/Esc U 撤回 五. 文件保存与退出5.1 Ctrl …...
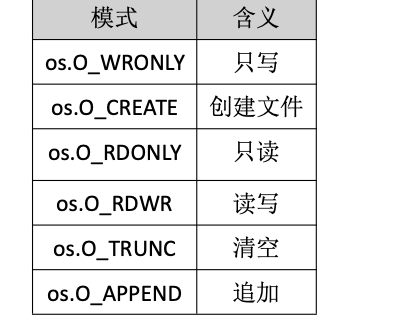
Golang——9、反射和文件操作
反射和文件操作 1、反射1.1、reflect.TypeOf()获取任意值的类型对象1.2、reflect.ValueOf()1.3、结构体反射 2、文件操作2.1、os.Open()打开文件2.2、方式一:使用Read()读取文件2.3、方式二:bufio读取文件2.4、方式三:os.ReadFile读取2.5、写…...
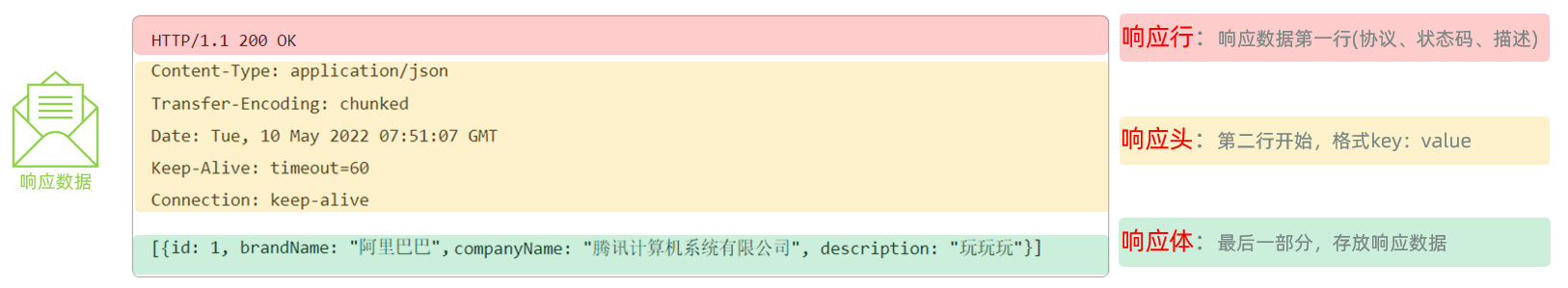
Web后端基础(基础知识)
BS架构:Browser/Server,浏览器/服务器架构模式。客户端只需要浏览器,应用程序的逻辑和数据都存储在服务端。 优点:维护方便缺点:体验一般 CS架构:Client/Server,客户端/服务器架构模式。需要单独…...
