WNN 多模态整合 | Seurat 单细胞多组学整合流程
测试环境:CentOS7.9, R4.3.2, Seurat 4.4.0, SeuratObject 4.1.4
2024.10.23
# WNN
library(ggplot2)
library(dplyr)
library(patchwork)
1. 导入数据
(1). load counts of RNA and protein
dyn.load('/home/wangjl/.local/lib/libhdf5_hl.so.100')
library(hdf5r)library(Seurat)
dat=Read10X_h5("/datapool/wangjl/others/hanlu/raw/GSE210079/GSM6459763_32-3mo_raw_feature_bc_matrix.h5")
str(dat)
names(dat) #"Gene Expression" "Antibody Capture" #两个矩阵:RNA和 55个蛋白str(dat$`Gene Expression`)
dat$`Gene Expression`[1:4, 1:5]# make sure cell id are the same
all.equal(colnames(dat[["Gene Expression"]]), colnames(dat[["Antibody Capture"]])) #T
(2). use RNA data to create Obj
scRNA=CreateSeuratObject(counts = dat$`Gene Expression`, project = "A1")
(3). add protein mat
# https://zhuanlan.zhihu.com/p/567253121
adt_assay <- CreateAssayObject(counts = dat$`Antibody Capture`)
scRNA[["ADT"]] <- adt_assay# (4). check
# protein names
rownames(scRNA[["ADT"]])# assays
Assays(scRNA) #"RNA" "ADT"# check default assay, or change default assay
DefaultAssay(scRNA) #"RNA"
2. 每个模态分别分析
要分别分析到PCA结束。
bm=scRNA## QC ====
bm #655671
bm[["percent.mt"]] <- PercentageFeatureSet(bm, pattern = "^MT-")# VlnPlot(bm, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)plot1 <- FeatureScatter(bm, feature1 = "nCount_RNA", feature2 = "percent.mt")
plot2 <- FeatureScatter(bm, feature1 = "nCount_RNA", feature2 = "nFeature_RNA")
(plot1 + geom_hline(yintercept = 10, linetype=2, color="red") ) + (plot2 + geom_hline(yintercept = c(300, 5000), linetype=2, color="red")) #fig1

Fig1
(1)Filter
bm <- subset(bm, subset = nFeature_RNA > 300 & nFeature_RNA < 5000 & percent.mt < 10)
bm #7837
(2)for RNA
DefaultAssay(bm) <- 'RNA'
bm <- NormalizeData(bm) %>% FindVariableFeatures(nfeatures = 3000) %>% ScaleData() %>% RunPCA(dims = 1:50)
DimPlot(bm, reduction = 'pca')
ElbowPlot(bm, ndims = 50) #fig2
(3)for protein
DefaultAssay(bm) <- 'ADT'
# we will use all ADT features for dimensional reduction
# we set a dimensional reduction name to avoid overwriting the
VariableFeatures(bm) <- rownames(bm[["ADT"]])
bm <- NormalizeData(bm, normalization.method = 'CLR', margin = 2) %>% ScaleData() %>% RunPCA(reduction.name = 'apca')
ElbowPlot(bm, ndims = 50, reduction = "apca") #fig2

Fig2
3. 整合模态
# Identify multimodal neighbors. These will be stored in the neighbors slot,
# and can be accessed using bm[['weighted.nn']] 加权最近邻
# The WNN graph can be accessed at bm[["wknn"]], 加权knn图
# and the SNN graph used for clustering at bm[["wsnn"]] 加权snn图
# Cell-specific modality weights can be accessed at bm$RNA.weight #模态的权重
bm2=bm
bm2 <- FindMultiModalNeighbors(bm,reduction.list = list("pca", "apca"),dims.list = list(1:30, 1:20)#modality.weight.name = c("RNA.weight", "ADT.weight")# 模态权重名字 要和 reduction.list 长度一致,否则会使用默认:assay + ".weight"
)
bm2@graphs |> names() #[1] "wknn" "wsnn"
4. 基于wnn的下游分析
(1)UMAP和细胞分群
bm2 <- RunUMAP(bm2, nn.name = "weighted.nn", reduction.name = "wnn.umap", reduction.key = "wnnUMAP_")
bm2 <- FindClusters(bm2, graph.name = "wsnn", algorithm = 1, resolution = 0.7, verbose = T)
#0.3,0.4,0.6 too small; 0.8 too large;p1=DimPlot(bm2, reduction = 'wnn.umap', label=T, group.by = 'wsnn_res.0.7') + ggtitle("WNN"); p1 #fig3

Fig3 (same as Fig8)
(2)模态权重:按cluster统计
head(bm3@meta.data)
VlnPlot(bm3, features = c("RNA.weight", "nFeature_RNA", "ADT.weight", "nFeature_ADT"), group.by = 'wsnn_res.0.7', sort = F, #是否排序pt.size = 0, ncol = 2) +NoLegend() #Fig3B# 每个细胞的2个模态中的权重和为1
all( abs((bm3@meta.data$RNA.weight + bm3@meta.data$ADT.weight) -1) < 1e-10) #T

Fig3B
5. 和单一模态的比较
bm3=bm2
DefaultAssay(bm3)="RNA" #RNA
DefaultAssay(bm3) #RNA
(1) 单模态UMAP
bm3 <- RunUMAP(bm3, reduction = 'pca', dims = 1:30, assay = 'RNA', reduction.name = 'rna.umap', reduction.key = 'rnaUMAP_')
bm3 <- RunUMAP(bm3, reduction = 'apca', dims = 1:20, assay = 'ADT', reduction.name = 'adt.umap', reduction.key = 'adtUMAP_')
bm3@reductions |> names() #[1] "pca" "apca" "wnn.umap" "rna.umap" "adt.umap"p2 <- DimPlot(bm3, reduction = 'rna.umap', #group.by = 'celltype.l2', label = TRUE, #label.size = 2.5,repel = TRUE) + ggtitle("RNA") + NoLegend()
p3 <- DimPlot(bm3, reduction = 'adt.umap', #group.by = 'celltype.l2', label = TRUE, #label.size = 2.5,repel = TRUE) + ggtitle("ADT")+ NoLegend()
p2 + p3 + p1 #Fig3if(0){
p4 <- FeaturePlot(bm3, features = c("adt_CD45RA","adt_CD14.1","adt_CD161"),reduction = 'wnn.umap', max.cutoff = 2, cols = c("lightgrey","darkgreen"), ncol = 3)
p5 <- FeaturePlot(bm3, features = c("rna_PTPRC", "rna_CD14", "rna_KLRB1"), reduction = 'wnn.umap', max.cutoff = 3, ncol = 3)
p4 / p5
}grep("CD45", bm3@assays$ADT@var.features, value=T) #"CD45RA" "CD45" "CD4.1" "CD45RO"
grep("FCGR3A", rownames(bm3@assays$RNA@counts), value=T)
FeatureScatter(bm3, feature1 = "adt_CD4.1", feature2 = "adt_CD8a")
FeatureScatter(bm3, feature1 = "adt_CD45", feature2 = "adt_CD8a") #Fig4

Fig4
#RNA UMAP
pC1=FeaturePlot(bm3, features = c("adt_CD45RA","adt_CD45RO", "adt_CD3","adt_CD4.1", "adt_CD8a", "adt_CD19.1"),reduction = 'rna.umap', max.cutoff = 2, cols = c("lightgrey","darkgreen"), ncol = 6) & NoLegend(); pC1
pC2=FeaturePlot(bm3, features = c("rna_PTPRC", "rna_CCR7", "rna_CD3D", "rna_CD4", "rna_CD8A", "rna_CD19"),reduction = 'rna.umap', max.cutoff = 2, cols = c("lightgrey","navy"), ncol = 6)& NoLegend(); pC2
pC1 / pC2 #Fig5

Fig5
#ADT UMAP
pC1=FeaturePlot(bm3, features = c("adt_CD45RA","adt_CD45RO", "adt_CD3","adt_CD4.1", "adt_CD8a", "adt_CD19.1"),reduction = 'adt.umap', max.cutoff = 2, cols = c("lightgrey","darkgreen"), ncol = 6) & NoLegend(); pC1
pC2=FeaturePlot(bm3, features = c("rna_PTPRC", "rna_CCR7", "rna_CD3D", "rna_CD4", "rna_CD8A", "rna_CD19"),reduction = 'adt.umap', max.cutoff = 2, cols = c("lightgrey","navy"), ncol = 6)& NoLegend(); pC2
pC1 / pC2 #Fig6

Fig6
# WNN
pC1=FeaturePlot(bm3, features = c("adt_CD45RA","adt_CD45RO", "adt_CD3","adt_CD4.1", "adt_CD8a", "adt_CD19.1"),reduction = 'wnn.umap', max.cutoff = 2, cols = c("lightgrey","darkgreen"), ncol = 6) & NoLegend(); pC1
pC2=FeaturePlot(bm3, features = c("rna_PTPRC", "rna_CCR7", "rna_CD3D", "rna_CD4", "rna_CD8A", "rna_CD19"),reduction = 'wnn.umap', max.cutoff = 2, cols = c("lightgrey","navy"), ncol = 6)& NoLegend(); pC2
pC1 / pC2 #Fig7

Fig7 效果似乎不好,CD4+和CD8+依旧不清晰。
也没有其他更优的参数可以调试。
也就是wnn不一定适合所有该类型(RNA + ADT)的样本。
(2) 单模态细胞聚类/cell cluster
DefaultAssay(bm3)="RNA"
bm3@graphs |> names() #[1] "wknn" "wsnn"
bm3 <- FindNeighbors(bm3, dims = 1:30, reduction = "pca")
bm3@graphs |> names() ##[1] "wknn" "wsnn" "RNA_nn" "RNA_snn"
bm3 <- FindClusters(bm3, graph.name = "RNA_snn", algorithm = 1, resolution = 0.5)
table(bm3@meta.data$RNA_snn_res.0.5)DefaultAssay(bm3)="ADT"
DefaultAssay(bm3) #ADT
bm3 <- FindNeighbors(bm3, dims = 1:20, reduction = "apca")
bm3@graphs |> names() #[1] "wknn" "wsnn" "RNA_nn" "RNA_snn" "ADT_nn" "ADT_snn"
bm3 <- FindClusters(bm3, graph.name = "ADT_snn", algorithm = 1, resolution = 0.5)
table(bm3@meta.data$ADT_snn_res.0.5)pB1 <- DimPlot(bm3, reduction = 'rna.umap', group.by = 'RNA_snn_res.0.5', label = TRUE, #label.size = 2.5,repel = F) + ggtitle("RNA umap & its cluster")
pB2 <- DimPlot(bm3, reduction = 'adt.umap', group.by = 'ADT_snn_res.0.5', label = TRUE, #label.size = 2.5,repel = F) + ggtitle("ADT umap & its cluster")
pB3=DimPlot(bm3, reduction = 'wnn.umap', group.by = 'wsnn_res.0.7', label=T) + ggtitle("WNN");
pB1 + pB2 + pB3 #Fig8

Fig8 (same as Fig3B)
- https://satijalab.org/seurat/articles/weighted_nearest_neighbor_analysis
相关文章:

WNN 多模态整合 | Seurat 单细胞多组学整合流程
测试环境:CentOS7.9, R4.3.2, Seurat 4.4.0, SeuratObject 4.1.4 2024.10.23 # WNN library(ggplot2) library(dplyr) library(patchwork)1. 导入数据 (1). load counts of RNA and protein dyn.load(/home/wangjl/.local/lib/libhdf5_hl.so.100) library(hdf5r)…...

【Linux】磁盘文件系统(inode)、软硬链接
文章目录 1. 认识磁盘1.1 磁盘的物理结构1.2 磁盘的逻辑结构 2. 引入文件系统2.1 EXT系列文件系统的分区结构2.2 inode 3. 软硬链接3.1 软链接3.2 硬链接 在讲过了内存文件系统后,我们可以知道文件分为两种: 打开的文件(内存中)未…...
网安加·百家讲坛 | 徐一丁:金融机构网络安全合规浅析
作者简介:徐一丁,北京小西牛等保软件有限公司解决方案部总监,网络安全高级顾问。2000年开始从事网络安全工作,主要领域为网络安全法规标准研究、金融行业安全咨询与解决方案设计、信息科技风险管理评估等。对国家网络安全法规标准…...
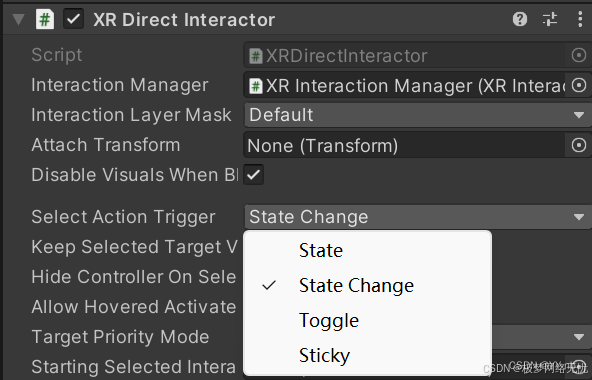
九、pico+Unity交互开发——触碰抓取
一、VR交互的类型 Hover(悬停) 定义:发起交互的对象停留在可交互对象的交互区域。例如,当手触摸到物品表面(可交互区域)时,视为触发了Hover。 Grab(抓取) 概念ÿ…...

老机MicroServer Gen8再玩 OCP万兆光口+IT直通
手上有一台放了很久的GEN8微型服务器,放了很多年,具体什么时候买的我居然已经记不清了 只记得开始装修的时候搬家出去就没用了,结果搬出去有了第1个孩子,孩子小的时候也没时间折腾,等孩子大一点的时候,又有…...

jmeter 从多个固定字符串中随机取一个值的方法
1、先新增用户参数,将固定值设置为不同的变量 2、使用下面的函数,调用这写变量 ${__RandomFromMultipleVars(noticeType1|noticeType2|noticeType3|noticeType4|noticeType5)} 3、每次请求就是随机取的值了...

priority_queue (优先级队列的使用和模拟实现)
使用 priority_queue 优先级队列与 stack 和 queue 一样,也是一个容器适配器,其底层通过 vector 来实现的。与 stack 和 queue 不同的是,它的第一个元素总是它所包含的元素中最大或最小的一个。 也就是说,优先级队列就是数据结…...
VisionPro 手部骨骼跟踪 Skeletal Hand Tracking 虚拟首饰
骨骼手部跟踪由XR Hands Package中的Hand Subsystem提供。使用场景中的Hand Visualizer组件,用户可以显示玩家手部的蒙皮网格或每个关节的几何图形,以及用于基于手部物理交互的物理对象。用户可以直接针对Hand Subsystem编写 C# 脚本,以推断骨…...
class 9: vue.js 3 组件化基础(2)父子组件间通信
目录 父子组件之间的相互通信父组件传递数据给子组件Prop为字符串类型的数组Prop为对象类型 子组件传递数据给父组件 父子组件之间的相互通信 开发过程中,我们通常会将一个页面拆分成多个组件,然后将这些组件通过组合或者嵌套的方式构建页面。组件的嵌套…...

Laravel|Lumen项目配置信息config原理
介绍 Laravel 框架的所有配置文件都保存在 config 目录中。每个选项都有说明,你可随时查看这些文件并熟悉都有哪些配置选项可供你使用。 使用 您可以在应用程序的任何位置使用全局 config 辅助函数轻松访问配置值。 可以使用“点”语法访问配置值,其中…...

2024系统分析师考试---论区块链技术及其应用
试题三论区块链技术及其应用 区块链作为一种分布式记账技术,目前已经被应用到了资产管理、物联网、医疗管理、政务监管等多个领域,从网络层面来讲,区块链是一个对等网络(Peer to Peer,P2P),网络中的节点地位对等,每个节点都保存完整的账本数据,系统的运行不依赖中心化节…...
)
为您的 Raspberry Pi 项目选择正确的实时操作系统(RTOS)
在嵌入式系统设计中,实时操作系统(RTOS)的选择对于确保项目的实时性能和可靠性至关重要。Raspberry Pi,尤其是其最新的RP2040微控制器,为开发者提供了一个功能强大的平台来实现各种实时应用。本文将探讨如何为您的Rasp…...

鸿蒙应用的Tabs 组件怎么使用
鸿蒙应用中的Tabs组件是一个用于通过页签进行内容视图切换的容器组件,每个页签对应一个内容视图。以下是Tabs组件的使用方法: 一、基本结构 Tabs组件的页面组成包含两个部分,分别是TabContent和TabBar。TabContent是内容页,TabB…...

第四天 文件操作与异常处理
在Python中,文件操作是处理输入输出的基本操作之一,而异常处理则用于管理潜在的错误情况,确保程序的健壮性和稳定性。下面将介绍Python中的文件操作和异常处理的基本用法。 文件操作 打开文件 使用内置的 open() 函数可以打开一个文件&…...

【密码分析学 笔记】ch3 3.1 差分分析
ch3 分组密码的差分分析和相关分析方法 3.1 差分分析 评估分组密码安全性通用方法可用于杂凑函数和流密码安全性 预备知识: 迭代性分组密码(分组密码一般结构)简化版本 mini-AES CipherFour算法 3.1.1 差分分析原理 现象:密…...

Go:strings包的基本使用
文章目录 string前缀和后缀字符串包含判断子字符串或字符在父字符串中出现的位置字符串替换统计字符串出现次数重复字符串修改字符串大小写修剪字符串分割字符串拼接 slice 到字符串 strconv 本篇主要总结的是go中的string包的一些函数的操作讲解 string 在各个语言中&#x…...

uniapp,获取头部高度
头部自定义时候,设置获取安全区域,可以用 uni.getSystemInfoSync();接口。 <view class"statusBar" :style"{height:statusBarHeightpx}"> let SYSuni.getSystemInfoSync(); let statusBarHeightref(SYS.statusBarHeight) …...

开发面试题-更新中...
探迹科技(腾讯面试官) 1.了不了解循环屏障 2.对于java中的线程冲突有多少了解(我要算1加到1亿) 3.mysql调优怎么调(我跟他讲了explain) 4.type中ref,range,const的区别 5.我有1亿的数据量&…...

【Jmeter】jmeter指定jdk版本启动
背景: 因权限问题,不能修改操作系统的环境变量或者因jmeter启动加载的默认jdk8版本低,需要指定jdk XX版本启动Jmeter 解决办法: 进入jmeter bin目录选择jmeter.bat,记事本编辑jmeter.bat, 在最前面添加 set MINIMAL_…...

数据处理利器:图片识别转Excel表格让数据录入变简单
在现代职场中,手动录入数据是一个耗时且容易出错的过程。无论是纸质文件、照片还是截图,繁琐的输入常常让人感到头疼。如何高效地将这些信息转化为电子表格,是许多职场人士面临的挑战。 为了解决这一问题,我们推出了图片识别转Exc…...
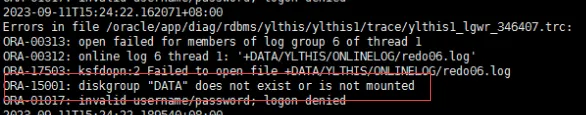
19c补丁后oracle属主变化,导致不能识别磁盘组
补丁后服务器重启,数据库再次无法启动 ORA01017: invalid username/password; logon denied Oracle 19c 在打上 19.23 或以上补丁版本后,存在与用户组权限相关的问题。具体表现为,Oracle 实例的运行用户(oracle)和集…...

CTF show Web 红包题第六弹
提示 1.不是SQL注入 2.需要找关键源码 思路 进入页面发现是一个登录框,很难让人不联想到SQL注入,但提示都说了不是SQL注入,所以就不往这方面想了 先查看一下网页源码,发现一段JavaScript代码,有一个关键类ctfs…...

SciencePlots——绘制论文中的图片
文章目录 安装一、风格二、1 资源 安装 # 安装最新版 pip install githttps://github.com/garrettj403/SciencePlots.git# 安装稳定版 pip install SciencePlots一、风格 简单好用的深度学习论文绘图专用工具包–Science Plot 二、 1 资源 论文绘图神器来了:一行…...

2025年能源电力系统与流体力学国际会议 (EPSFD 2025)
2025年能源电力系统与流体力学国际会议(EPSFD 2025)将于本年度在美丽的杭州盛大召开。作为全球能源、电力系统以及流体力学领域的顶级盛会,EPSFD 2025旨在为来自世界各地的科学家、工程师和研究人员提供一个展示最新研究成果、分享实践经验及…...

uni-app学习笔记二十二---使用vite.config.js全局导入常用依赖
在前面的练习中,每个页面需要使用ref,onShow等生命周期钩子函数时都需要像下面这样导入 import {onMounted, ref} from "vue" 如果不想每个页面都导入,需要使用node.js命令npm安装unplugin-auto-import npm install unplugin-au…...

连锁超市冷库节能解决方案:如何实现超市降本增效
在连锁超市冷库运营中,高能耗、设备损耗快、人工管理低效等问题长期困扰企业。御控冷库节能解决方案通过智能控制化霜、按需化霜、实时监控、故障诊断、自动预警、远程控制开关六大核心技术,实现年省电费15%-60%,且不改动原有装备、安装快捷、…...
)
python爬虫:Newspaper3k 的详细使用(好用的新闻网站文章抓取和解析的Python库)
更多内容请见: 爬虫和逆向教程-专栏介绍和目录 文章目录 一、Newspaper3k 概述1.1 Newspaper3k 介绍1.2 主要功能1.3 典型应用场景1.4 安装二、基本用法2.2 提取单篇文章的内容2.2 处理多篇文档三、高级选项3.1 自定义配置3.2 分析文章情感四、实战案例4.1 构建新闻摘要聚合器…...
 自用)
css3笔记 (1) 自用
outline: none 用于移除元素获得焦点时默认的轮廓线 broder:0 用于移除边框 font-size:0 用于设置字体不显示 list-style: none 消除<li> 标签默认样式 margin: xx auto 版心居中 width:100% 通栏 vertical-align 作用于行内元素 / 表格单元格ÿ…...
保姆级教程:在无网络无显卡的Windows电脑的vscode本地部署deepseek
文章目录 1 前言2 部署流程2.1 准备工作2.2 Ollama2.2.1 使用有网络的电脑下载Ollama2.2.2 安装Ollama(有网络的电脑)2.2.3 安装Ollama(无网络的电脑)2.2.4 安装验证2.2.5 修改大模型安装位置2.2.6 下载Deepseek模型 2.3 将deepse…...
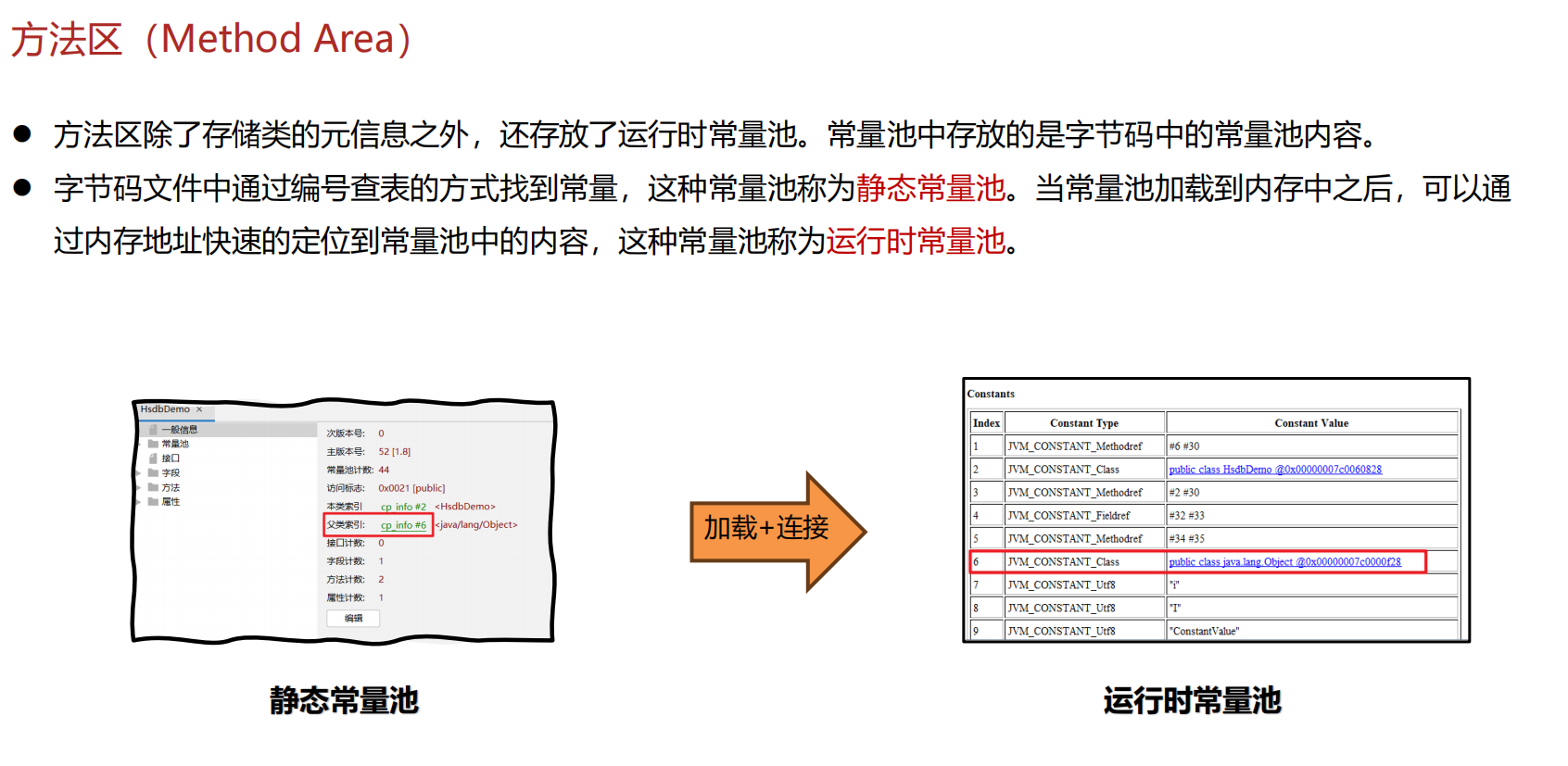
JVM 内存结构 详解
内存结构 运行时数据区: Java虚拟机在运行Java程序过程中管理的内存区域。 程序计数器: 线程私有,程序控制流的指示器,分支、循环、跳转、异常处理、线程恢复等基础功能都依赖这个计数器完成。 每个线程都有一个程序计数…...
