【中药网络药理学】筛选细胞衰老和预后相关基因(附分类代码和画图代码)
1、衰老相关基因
从HAGR和msigdb数据获取细胞衰老相关基因,将两者取交集后构建基因蛋白互作网络
HAGR数据库
该库本身提供了下载链接,我在下载后对其进行了清洗
msigdb数据库
以"aging"作为关键词,Search Filters中collection设置为"all collections",source species设置为"Homo sapiens",contributors设置为"all contributions"
在msigdb数据库中共得到56个衰老相关基因集
2、小细胞肝癌预后相关基因_TCGA数据库
library("survival")
library("survminer")
使用TCGA数据库通过TCGA-LIHC数据集(2023年1月,n=377),下载clinical(临床信息),点击Metadata(样本信息),cart(基因文件)[/data/TCGA数据库]获取转录组测序数据与临床数据,并排除了临床数据不完整的患者队列
我使用了Harmony软件包进行批次效应调整,
接下来,基于拟合多因素Cox回归和lasso回归,构建预后效果评价分类模型
以"stranded_first"作为特定基因的表达水平评价量,临床信息中选择vital_status:生存状态(例如,存活、死亡)和days_to_death:诊断后至死亡的天数,作为评价的临床指标,最后整合得到如下表格:
| 病例ID | 基因名 | 生存时间 | 结局 |
|---|---|---|---|
| ID1 | GeneA | 时间1 | 结果1 |
| ID2 | GeneB | 时间2 | 结果2 |
| ID3 | GeneC | 时间3 | 结果3 |
描述性分析
首先使用生存分析中的Cox比例风险模型建立一个风险评分系统,然后利用风险评分中位数对其进行分组,将其分为“高风险组”和”低风险组“,并绘制生存分析图(针对筛选出的基因)
# 生存分析
load("files/exp_clinical")install.packages("survival")
install.packages("survminer")
install.packages("glmnet")
install.packages("caret")library(survival)
library(survminer)
library(glmnet)
library(dplyr)
library(caret)# 绘制Kaplan-Meier曲线
exp_clinical <- as.data.frame(t(exp_clinical))
exp_clinical$time <- as.numeric(exp_clinical$time)
exp_clinical <- exp_clinical[!is.na(exp_clinical$time), ]# 观察小范围内数据框
View(exp_clinical[(1:50), (1:50)])
View(gene_data[(1:50), (1:50)])
View(normalized_data[(1:50), (1:50)])gene_data <- exp_clinical[(1:(nrow(exp_clinical)-5)), ]
clinical_data <- exp_clinical[((nrow(exp_clinical)-4) : nrow(exp_clinical)), ]# 选择至少在一个样本中表达量高于该基因90%分位数的基因
sapply(gene_data, class)gene_data[is.na(gene_data)] <- 0
gene_data[] <- lapply(gene_data, function(x) as.numeric(as.character(x)))gene_data_scale <- as.data.frame(scale(gene_data))normalize_minmax <- function(x) {return((x - min(x, na.rm = TRUE)) / (max(x, na.rm = TRUE) - min(x, na.rm = TRUE)))
}gene_data <- as.data.frame(lapply(gene_data_scale, normalize_minmax))names(gene_data) <- names(clinical_data)
data <- rbind(clinical_data, gene_data)
data <- as.data.frame(t(data))rownames(data)[6:nrow(data)] <- rownames(exp_clinical)[1:(nrow(exp_clinical)-5)]
save(data, file = "files/gene_clinical_data")# 做相关性分析
load("files/gene_clinical_data")View(data[(1:50), (1:50)])rm(list = ls())load("files/exp_clinical")
load("files/agging_HCC_tcm_gene")library(ggplot2)
library(ggpubr)
library(survival)
library(survminer)
library(glmnet)
library(mice)
library(dplyr)
library(car)
library(randomForestSRC)
library(caret)## 预处理
rownames(exp_clinical) <- data_rownames
exp_clinical <- replace(exp_clinical, is.na(exp_clinical), 0)
temp <- as.data.frame(t(exp_clinical))temp <- data.frame(lapply(temp, as.numeric))
temp <- replace(temp, is.na(temp), 0)colnames(temp) <- data_rownames
colnames(temp)gene_data <- as.data.frame(temp[, unlist(aging_HCC_tcm_gene)])
temp_gene_data <- as.data.frame(lapply(gene_data, as.numeric))str(temp_gene_data)temp_gene_data <- scale(temp_gene_data)
gene_data <- as.data.frame(temp_gene_data)
# temp <- scale(gene_data)rownames(gene_data) <- t(colnames(exp_clinical))exp_clinical <- as.data.frame(t(exp_clinical))
clinical_data <- as.data.frame(cbind(exp_clinical$time, exp_clinical$flag))colnames(clinical_data) <- c("time", "flag")clinical_data <- as.data.frame(lapply(clinical_data, as.numeric))
clinical_data[is.na(clinical_data)] <- 0sum(clinical_data$flag)rownames(clinical_data) <- rownames(gene_data)rows_to_remove <- rownames(clinical_data[clinical_data$time == 0, ])gene_data <- gene_data[!rownames(gene_data) %in% rows_to_remove, ]
clinical_data <- clinical_data[!rownames(clinical_data) %in% rows_to_remove, ]
# clinical_data <- clinical_data[!rownames(clinical_data) %in% rows_to_remove, ]# rownames(gene_data) <- t(colnames(exp_clinical))# clinical_data <- as.matrix(temp[, c("time", "flag")])
# clinical_data <- as.data.frame(clinical_data)
#
# clinical_data <- as.data.frame(lapply(clinical_data, as.numeric))
# sum(clinical_data$flag)
#
data <- cbind(clinical_data, gene_data)
# # temp_data <- cbind(clinical_data, temp)
#
# sum(is.na(clinical_data))
# sum(is.na(gene_data))# gene_data <- preProcess(gene_data, method = "range")save(data, gene_data, clinical_data, file = "files/surv_pre_data")## 风险回归
rm(list = ls())
load("files/surv_pre_data")str(clinical_data)
str(gene_data)custom_control <- coxph.control(eps = 1e-09, iter.max = 10000, toler.chol = 1e-10)surv_model <- coxph(Surv(time, flag) ~ ., data = data, control = custom_control)# temp_surv_model <- coxph(
# Surv(time, flag) ~ .,
# data = temp_data,
# control = coxph.control(iter.max = 10000)
#)surv_model$coefficients# surv_model <- temp_surv_modelcoefficients <- as.data.frame(surv_model$coefficients)
coefficients[is.na(coefficients)] <- 0
coefficientsgene_data <- data.frame(gene_data)
gene_coef_data <- rbind(gene_data, t(coefficients))
rownames(gene_coef_data)[nrow(gene_coef_data)] <- c("coef")surv_results <- sweep(gene_data, 2, t(coefficients), `*`)
surv_row_sums <- rowSums(surv_results)surv_row_sums[is.na(surv_row_sums)] <- 0
surv_row_sumssurv_median_value <- median(surv_row_sums)
surv_median_value# 分类并添加分组
risk_group <- ifelse(surv_row_sums > surv_median_value,"High Risk","Low Risk")
surv_results$risk_group <- risk_group
final_surv_results <- surv_results[, !apply(surv_results, 2,function(x) all(x == 0))]save(data, risk_group,gene_data, clinical_data, coefficients, final_surv_results, file = "files/surv_final_data")# 绘制生存曲线
rm(list = ls())load("files/surv_final_data")colnames(final_surv_results)temp_data <- data
temp_data$time <- ceiling(temp_data$time/30)
# temp_data <- lapply(temp_data, as.numeric)# 剔除大于5年的
rows_to_remove <- rownames(temp_data[temp_data$time > 60, ])
temp_data <- temp_data[!rownames(temp_data) %in% rows_to_remove, ]
clinical_data <- clinical_data[!rownames(clinical_data) %in% rows_to_remove, ]
risk_group <- as.data.frame(risk_group)
risk_group <- risk_group[!rownames(risk_group) %in% rows_to_remove, ]temp_surv_obj <- Surv(temp_data$time, temp_data$flag)
temp_data <- cbind(temp_data, risk_group)
temp_surv_fit <- survfit(temp_surv_obj ~ risk_group, data = temp_data)
temp_max <- max(temp_data$time)
ggsurvplot(temp_surv_fit, data = temp_data,risk.table = TRUE, # 显示风险表pval = TRUE, # 显示P值conf.int = TRUE, # 显示置信区间xlim = c(0, temp_max), # 可选:设置X轴的范围xlab = "Times(months)", # 设置X轴标签ylab = "Survival probability", # 设置Y轴标签
)coefficients$gene_name <- rownames(coefficients)
colnames(coefficients)[1] <- c("coef")
writexl::write_xlsx(coefficients, "data/washed_data/coef.xlsx")surv_obj <- Surv(data$time, data$flag)
surv_fit <- survfit(surv_obj ~ risk_group, data = data)
max <- max(data$time)
ggsurvplot(surv_fit, data = final_surv_results,risk.table = TRUE, # 显示风险表pval = TRUE, # 显示P值conf.int = TRUE, # 显示置信区间xlim = c(0, max), # 可选:设置X轴的范围xlab = "Times(days)", # 设置X轴标签ylab = "Survival probability", # 设置Y轴标签title = "Kaplan-Meier 生存曲线" # 设置图标题
)
相关文章:
)
【中药网络药理学】筛选细胞衰老和预后相关基因(附分类代码和画图代码)
1、衰老相关基因 从HAGR和msigdb数据获取细胞衰老相关基因,将两者取交集后构建基因蛋白互作网络 HAGR数据库 该库本身提供了下载链接,我在下载后对其进行了清洗 msigdb数据库 以"aging"作为关键词,Search Filters中collection…...

华为的流程体系
缘由 2010年,华为销售额为1850亿元,其中国际市场占65%,净利润238亿元。当时,公司员工达11万人,公司处理合同达5万多个,290万个订单,大量的工作是手工处理,没有统一的流程支持&#…...

算法——长度最小的子数组209 对比代码随想录题解中对于result取值为Integer.MAX_VALUE的思考
具体解题过程可看代码随想录,我主要是对于为什么result也就是子数组和初始化要为Integer.MAX_VALUE有一个疑惑,为什么不是其他值,经过思考后我发现: 情况一:如果result为负数的话是不符合数组长度取值的一个规范的。 情况二&…...

图像处理案例03
HOGSVM数字识别 1 . 步骤2 . 代码 1 . 步骤 读入数据,把数据划分为训练集和测试集用hog提取特征用SVM训练数据测试、评价模型保存模型加载模型,应用模型 2 . 代码 import os import cv2 import sklearn import numpy as np from skimage.feature impo…...

【Kubernetes】k8s集群中kubectl的陈述式资源管理
目录 一.k8s集群资源管理方式分类 1.陈述式资源管理方式 2.声明式资源管理方式 二.陈述式资源管理方法 三.kubectl命令 四.项目生命周期 1.创建 kubectl create命令 2.发布 kubectl expose命令 3.更新 kubectl set 4.回滚 kubectl rollout 5.删除 k…...

串---顺序串实现
顺序串详解 本文档将详细介绍顺序串的基本概念、实现原理及其在 C 语言中的具体应用。通过本指南,读者将了解如何使用顺序串进行各种字符串操作。 1. 什么是顺序串? 顺序串是一种用于存储字符串的数据结构,它使用一组连续的内存空间来保存…...

吴恩达机器学习WEEK2
COURSE1 WEEK2 多维特征 在线性回归中,往往特征不止一个,而是具有多维特征 例如,在预测房价的例子中,我们知道更多的信息: x 1 x_1 x1:房屋的面积 x 2 x_2 x2:卧室的数目 x 3 x_3 x3&a…...

yield and generator in python
首先,假设大家都对于pytyhon的List comprehension的使用有了一定经验(它可以用于list,set,和dict哦) 不熟悉的参考介绍: Comprehending Python’s Comprehensions – dbader.org generator generator是哦…...

spring原理(自学第六天)
Aware 接口及 InitializingBean 接口 今天将会学到Aware 接口及 InitializingBean 接口 我们可以先了解他们的作用: 1. Aware 接口用于注入一些与容器相关信息, 例如 a. BeanNameAware 注入 bean 的名字 b. BeanFactoryAware 注入…...
案例分享—国外优秀ui设计作品赏析
国外UI设计创意迭出,融合多元文化元素,以极简风搭配动态交互,打造沉浸式体验,色彩运用大胆前卫,引领界面设计新风尚 同时注重用户体验的深度挖掘,通过个性化定制与智能算法结合,让界面不仅美观且…...

【C++】简约与清晰的编程艺术
C编程的艺术:简约与清晰的实践之道 一、基础之美:基本类型与数据结构的力量二、函数与库类的艺术三、简约与清晰的实践之道 在C这一既古老又充满活力的编程语言世界里,程序员们常常面临着一个重要的选择:是追求代码的极致抽象与封…...

java之WIFI信号模块
开发步骤分为以下几点: 1.在 AndroidManifest 中声明相关权限(网络和文件读写权限) 声明权限: <uses-permission android:name"android.permission.ACCESS_WIFI_STATE" /> <uses-permission android:name"android.…...

Mybatis面试
Mybatis 面试 1、Mybatis 的执行流程是什么? 1、读取MyBatis配置文件:mybatis-config.xml 加载运行环境 和 映射文件 2、构造会话工厂 SqlSessionFactory (全局只有一个) 3、会话工厂创建SqlSession对象(项目与数据…...
)
Centos 8系统xfs文件系统类型进行扩容缩容 (LVM)
Centos 8系统xfs文件系统类型进行扩容缩容 (LVM),xfs分区类型是不支持正常缩容,只能强制缩容 1.磁盘情况:2.缩容home分区1.备份home数据:2.查找使用 /home 的进程:3.终止这些进程:4.卸载 /home …...

C语言基础知识之函数指针和指针函数
函数指针和指针函数 函数指针和指针函数指向函数的指针返回指针值的函数指针函数和函数指针的区别 问题1_1代码1_1结果1_1 函数指针和指针函数 指向函数的指针 用函数指针变量调用函数 可以用指针变量指向整型变量、字符串、数组,也可以指向一个函数。一个…...

【Unity】web gl inputFied 中文输入,同时支持TextMeshInputFied,支持全屏
同时支持TextMeshInputFied,支持全屏。 使用github包【WebGLInput】:https://github.com/kou-yeung/WebGLInput 需要资源的在这里也可以下载 https://download.csdn.net/download/weixin_46472622/89600795 用于unity web gl 中文输入,只需…...

vue3+vite全局引入less变量和函数
需要在vite配置 plugins: [css: {preprocessorOptions: {less: {additionalData: import "./src/styles/variables.module.less"; import "./src/views/Visualization/component/ViewportCom/px2viewport.less";,javascriptEnabled: true}}}, ]多个文件按…...

H81002S 1.7mm网络变压器:BMS汽车蓝牙接收器中的超薄共模电感科技
华强盛导读:在当今这个日新月异的汽车科技领域,每一处细节都蕴含着创新与突破。作为电动汽车心脏的电池管理系统(BMS),其高效稳定的运行不仅关乎续航与安全,更是智能化驾驶体验的基石。而在这背后ÿ…...

C语言.回调函数
回调函数 回调函数也是一个函数。与一般函数直接调用区别在于,使用回调函数的过程,是一个函数将另一个函数作为参数调用。而被用来调用的那个函数,就是回调函数。 回调函数就是一个通过函数指针调用的函数。如果你把函数的指针(地…...

《从零开始:使用Python构建简单Web爬虫》
前言 随着互联网信息的爆炸性增长,如何高效地获取和处理这些数据变得越来越重要。Web爬虫作为一种自动化工具,可以帮助我们快速抓取所需的网页内容。本文将介绍如何使用Python编写一个简单的Web爬虫,并通过实例演示其基本用法。 准备工作 …...
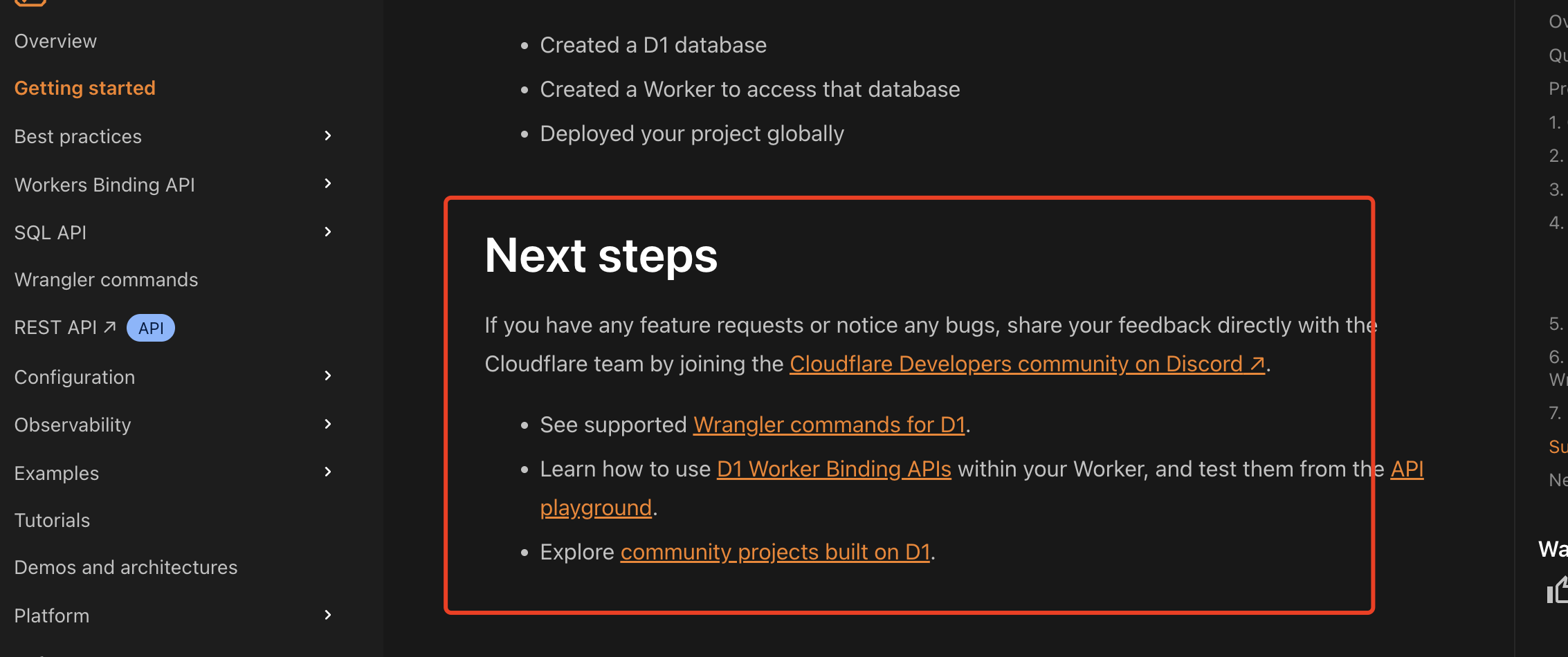
通过Wrangler CLI在worker中创建数据库和表
官方使用文档:Getting started Cloudflare D1 docs 创建数据库 在命令行中执行完成之后,会在本地和远程创建数据库: npx wranglerlatest d1 create prod-d1-tutorial 在cf中就可以看到数据库: 现在,您的Cloudfla…...
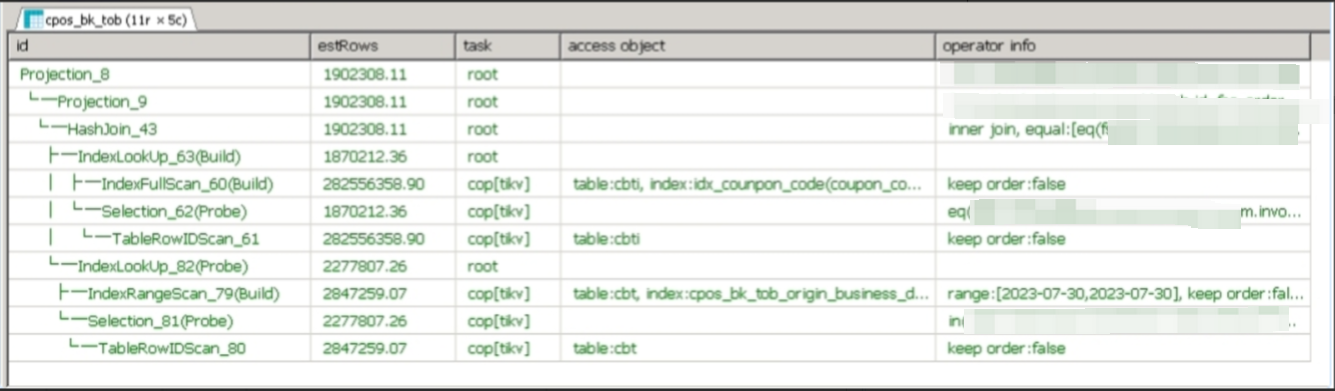
【入坑系列】TiDB 强制索引在不同库下不生效问题
文章目录 背景SQL 优化情况线上SQL运行情况分析怀疑1:执行计划绑定问题?尝试:SHOW WARNINGS 查看警告探索 TiDB 的 USE_INDEX 写法Hint 不生效问题排查解决参考背景 项目中使用 TiDB 数据库,并对 SQL 进行优化了,添加了强制索引。 UAT 环境已经生效,但 PROD 环境强制索…...

电脑插入多块移动硬盘后经常出现卡顿和蓝屏
当电脑在插入多块移动硬盘后频繁出现卡顿和蓝屏问题时,可能涉及硬件资源冲突、驱动兼容性、供电不足或系统设置等多方面原因。以下是逐步排查和解决方案: 1. 检查电源供电问题 问题原因:多块移动硬盘同时运行可能导致USB接口供电不足&#x…...

Nginx server_name 配置说明
Nginx 是一个高性能的反向代理和负载均衡服务器,其核心配置之一是 server 块中的 server_name 指令。server_name 决定了 Nginx 如何根据客户端请求的 Host 头匹配对应的虚拟主机(Virtual Host)。 1. 简介 Nginx 使用 server_name 指令来确定…...

Spring Boot面试题精选汇总
🤟致敬读者 🟩感谢阅读🟦笑口常开🟪生日快乐⬛早点睡觉 📘博主相关 🟧博主信息🟨博客首页🟫专栏推荐🟥活动信息 文章目录 Spring Boot面试题精选汇总⚙️ **一、核心概…...

Android Bitmap治理全解析:从加载优化到泄漏防控的全生命周期管理
引言 Bitmap(位图)是Android应用内存占用的“头号杀手”。一张1080P(1920x1080)的图片以ARGB_8888格式加载时,内存占用高达8MB(192010804字节)。据统计,超过60%的应用OOM崩溃与Bitm…...

Spring是如何解决Bean的循环依赖:三级缓存机制
1、什么是 Bean 的循环依赖 在 Spring框架中,Bean 的循环依赖是指多个 Bean 之间互相持有对方引用,形成闭环依赖关系的现象。 多个 Bean 的依赖关系构成环形链路,例如: 双向依赖:Bean A 依赖 Bean B,同时 Bean B 也依赖 Bean A(A↔B)。链条循环: Bean A → Bean…...
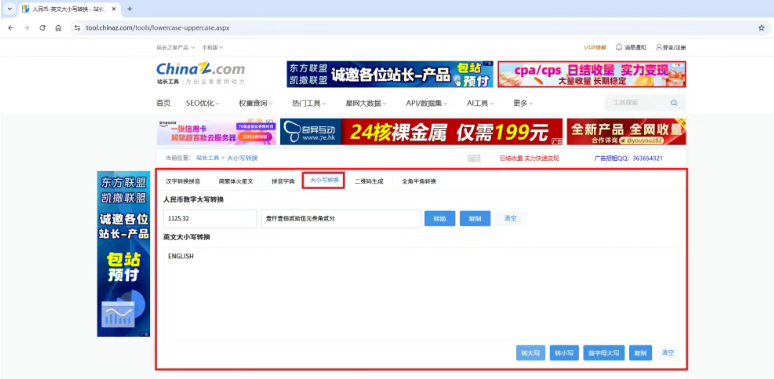
【分享】推荐一些办公小工具
1、PDF 在线转换 https://smallpdf.com/cn/pdf-tools 推荐理由:大部分的转换软件需要收费,要么功能不齐全,而开会员又用不了几次浪费钱,借用别人的又不安全。 这个网站它不需要登录或下载安装。而且提供的免费功能就能满足日常…...
【笔记】WSL 中 Rust 安装与测试完整记录
#工作记录 WSL 中 Rust 安装与测试完整记录 1. 运行环境 系统:Ubuntu 24.04 LTS (WSL2)架构:x86_64 (GNU/Linux)Rust 版本:rustc 1.87.0 (2025-05-09)Cargo 版本:cargo 1.87.0 (2025-05-06) 2. 安装 Rust 2.1 使用 Rust 官方安…...
)
C++课设:简易日历程序(支持传统节假日 + 二十四节气 + 个人纪念日管理)
名人说:路漫漫其修远兮,吾将上下而求索。—— 屈原《离骚》 创作者:Code_流苏(CSDN)(一个喜欢古诗词和编程的Coder😊) 专栏介绍:《编程项目实战》 目录 一、为什么要开发一个日历程序?1. 深入理解时间算法2. 练习面向对象设计3. 学习数据结构应用二、核心算法深度解析…...
